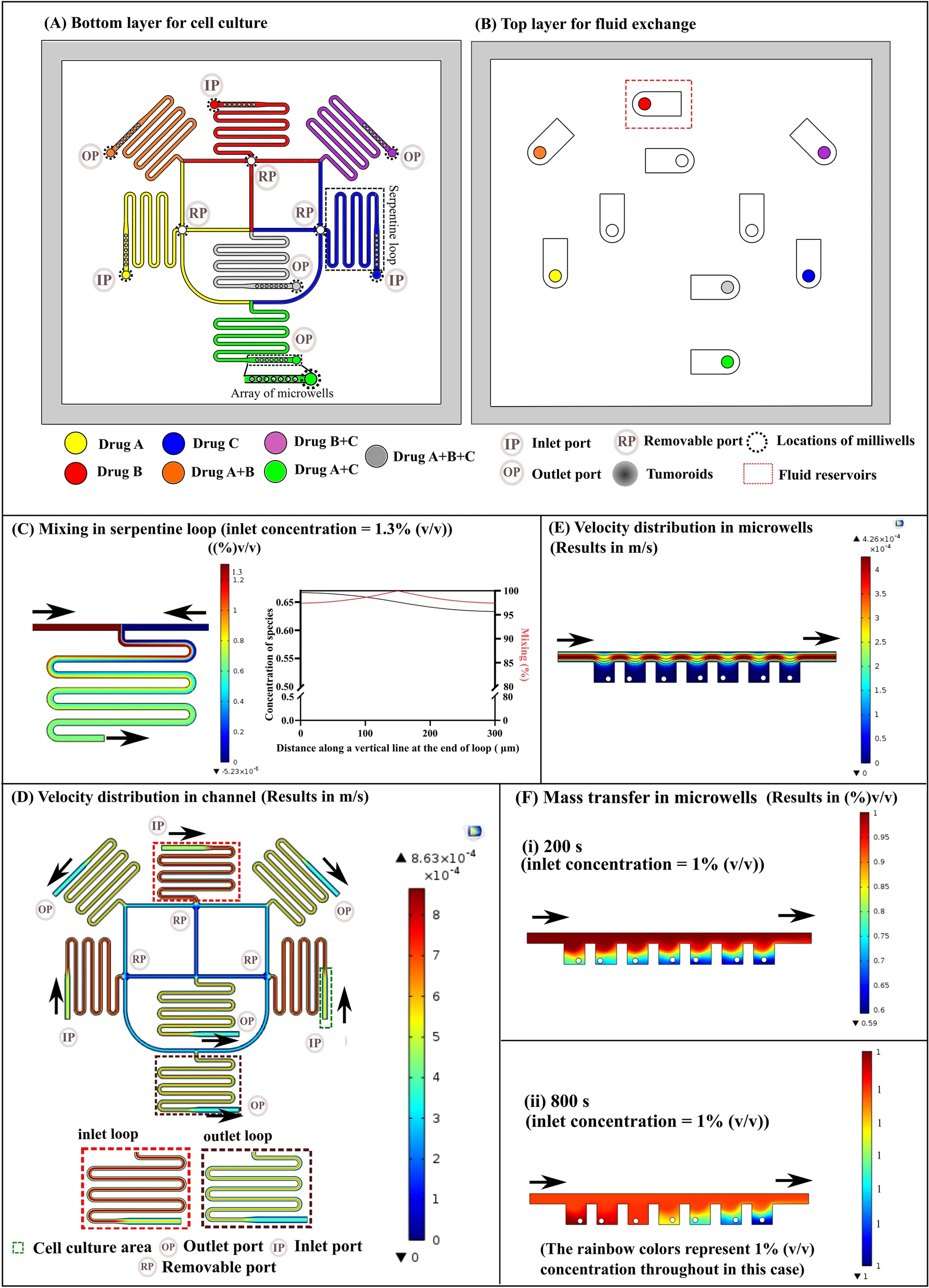
一种在单个芯片中进行高密度的三维培养、可控刺激和观察的微流控平台。
Introduction
三维(3D)培养可以调节单层(2D)培养中明显改变的许多重要功能。然而维持和操纵细胞球的固有困难阻碍了对细胞行为的高通量、定量测量。获得此类数据依赖于:
- 流式细胞术,但会失去三维培养中表型与细胞位置之间关系的所有信息。
- 新开发的显微镜与图像分析方法,但通量有限。
目前生产细胞球的方法:
- 传统的批量方法,如转瓶法或低附着力板。可以产生大量的细胞球,但对其大小分布和培养环境的控制有限。
- 利用微细加工提供一种自下而上的方法,在受控条件下将细胞聚集在一起(如AggreWell™板等)。但调节培养环境是劳动密集型,如果不使用复杂的自动化系统就无法实现并行化。
这些局限促使人们在微流体通道内实施三维培养方法,以弥补现有方法的不足。目前已在微通道底板上使用微孔进行了长期细胞球培养和观察,这些孔可进行可控灌注的多条件刺激和原位分析。但是,这些平台仅针对少量的细胞球进行了演示,并且分析仍然仅限于测量平均行为。相比之下,液滴方法提供了一种可扩展的方式来封装和限制样品,同时提供了多种操作工具。
在这种情况下,强烈需要用于可控三维细胞培养的高功能平台。实际上下一代平台将理想地在单个设备中集成多种功能,包括:
- 细胞球的生产;
- 细胞球维持活性与生产状态;
- 对细胞球环境的控制和调节(如刺激/药物);
- 原位单细胞染色和观察;
- 选择性回收任何感兴趣的细胞球以进行进一步分析或培养。
在本文中,作者展示了如何利用多种液滴操作技术将液滴微流控扩展到芯片上以提供高密度三维培养,并将其与液滴凝胶化结合起来以进行长期培养和单细胞观察。该平台不仅能进行群体规模的定量表征,还能产生数以千计的单个细胞球和细胞球内数十万个原位细胞的规模,同时允许提取单个细胞球进行进一步分析。这种通量和细节的同时增加揭示了细胞单位尺度上的异质性。通过严格控制芯片上的流体流动,作者证明了平台能够动态调节细胞球的生化微环境,并通过应用不同的药物暴露机制来区分细胞死亡动力学的变化。最终,该平台可以扩展到不同的共培养配置,为新的器官芯片方法铺平道路。
在本研究中,H4-II-EC3(大鼠肝癌细胞系)被用作肝细胞的模型。虽然这些细胞不表达原代肝细胞的所有解毒酶,但H4-II-EC3及其衍生物表达几种重要的细胞色素(CYP2B1 / 2、CYP2E和CYP3A)。并且H4-II-EC3表现出与HEPG2(一种成熟的人类肝癌,广泛用于毒理学研究)相似的药物化合物敏感性。此外,该平台还与原代祖细胞兼容,后者在组织工程中具有广泛的应用。
Droplet-microfluidic platform for the culture of spheroids
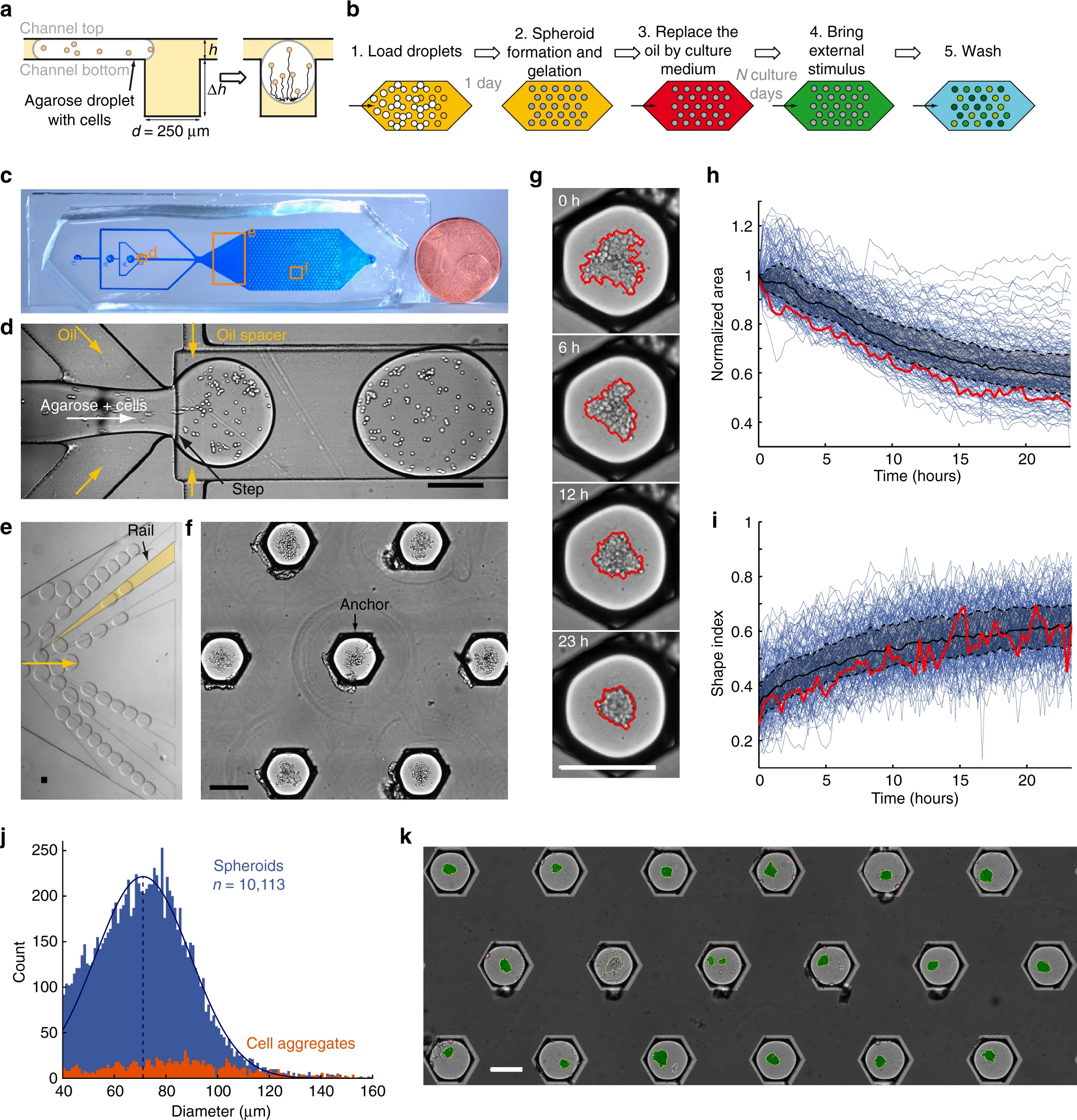
- (a)进入锚的液滴侧视示意图,由于表面能降低而被捕获。在停止油流后,封装的细胞沉积在液滴的底部并在培养一天后形成细胞球(h = 95 μm,Δh = 250 μm,D = 250 μm)。
- (b)先装入液滴,然后在细胞球形成后凝胶化。这样就可以用注入刺激物或抗体的培养基代替油。
- (c)PDMS微流控芯片照片。
- (d)液滴在增加了一个台阶的流动聚焦结合部形成。
- (e)发散导轨将液滴分散在培养室的整个宽度上。
- (f)一些代表性的含琼脂糖液滴的锚显示了细胞球形成前的沉降细胞。
- (g)一个锚内细胞球形成的时间间隔图。红线表示检测到的图案边缘。
- (h-i)细胞球形成的动力学:如图显示为152个细胞球的归一化面积(h)和形状指数(i)的变化。每条蓝线代表一个细胞球;红色实线代表g中的细胞球;黑色实线代表中位数;黑色虚线代表第一和第三分位数。
- (j)所有细胞球(蓝色,35个芯片,n = 10113)与细胞团(橙色,n = 1189)的尺寸分布。其中,细胞球定义为(D > 40 μm, ShI > 0.5);细胞团定义为(D > 40 μm, ShI < 0.5)。
- (k)对18个死活染色的充满细胞球的锚的图像分析。
形状指数(Shape index, ShI)计算方式为:
$$
ShI=\frac{4\pi A}{P^2}
$$
其中,A为面积,P为细胞周长。
Multiscale cytometry on chip
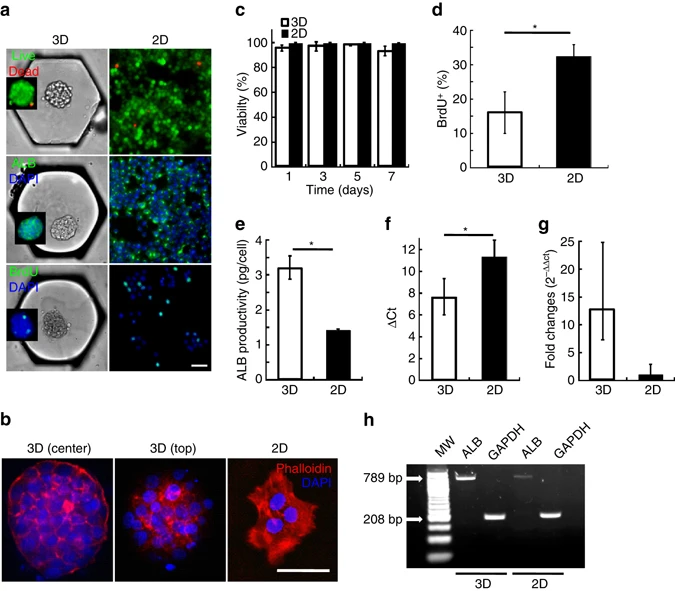
- (a)锚中细胞球与二维细胞的死活、ALB和BrdU染色的代表性图像。
- (b)肌动蛋白在芯片上的一个细胞球(两个垂直位置的共聚焦切片)和二维细胞上的组织。在细胞球上主要为皮质肌动蛋白的形式,二维细胞主要是胞质纤维的形式。
- (c)在7天的培养期间,芯片上细胞球和二维细胞的细胞活性演变。细胞活性均大于95%。
- (d)在7天的培养期间,芯片上细胞球和二维细胞的BrdU+细胞的百分比定量。细胞球中BrdU+细胞比例为二维细胞的一半。
- (e)第4天,通过酶联免疫吸附法(ELISA)测定上清,得到芯片上细胞球和二维细胞的白蛋白(ALB)产量。
- (f)RT-qPCR分析芯片上细胞球和二维细胞的ALB相对GADPH的表达(ΔCt)。
- (g)RT-qPCR分析芯片上细胞球和二维细胞的ALB表达(相对RNA表达)。
- (h)代表性凝胶电泳分析RT-PCR分析芯片上细胞球和二维细胞的ALB和GAPDH的表达。由此可见,在转录组水平,细胞球相比于二维细胞有约10倍的增加。
与二维细胞相比,细胞骨架组织的改变,增殖降低和ALB表达增加表明,在微流控平台上培养的H4-II-EC3具有高功能性肝细胞球的结构和表型特征。
有关RT-qPCR的计算,见此。
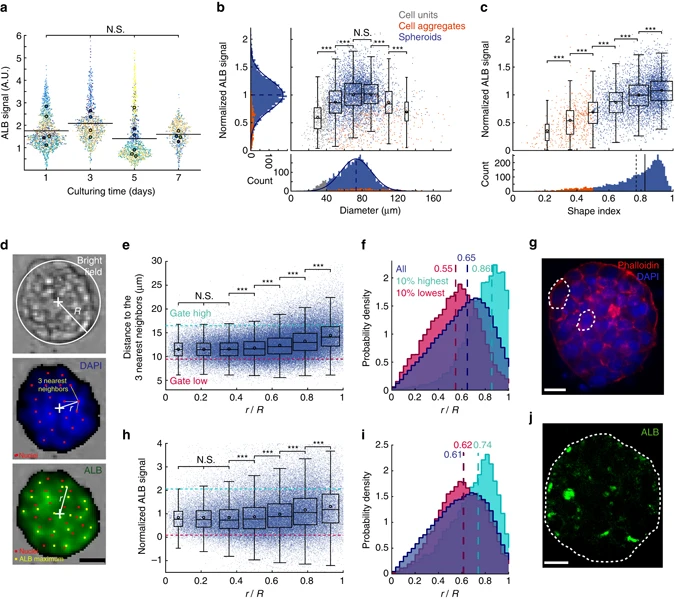
- (a)测量的细胞内ALB振幅。每个点代表一个细胞球;每个颜色代表一个芯片;每个芯片的平均值用一个颜色相同的大点表示;黑色实线代表各时间点的平均ALB信号。尽管细胞球群体没有显示出细胞内ALB信号随培养时间的显著变化,但每个实验中的单个细胞球显示出与其大小和形状相关的变化。
- (b-c)每个细胞球的归一化ALB信号与细胞球直径(b)和形状指数(c)的关系。通过将一个特定的球体ALB强度值除以芯片上ALB信号的平均值来进行归一化。通过这种方式,可以比较不同的芯片上细胞球的ALB产量。直方图显示了数据沿不同轴的分布。蓝色曲线为高斯拟合,蓝色虚线为平均值。更高的形状指数(0.8-1)显示了更高的细胞内ALB表达。这种产率对形状的依赖支持了功能细胞-细胞间相互作用的作用,可以促进细胞球形成和ALB的表达。
- (d)白光图(上),DAPI染色图(中),ALB染色图(下)。R为细胞球半径,r为细胞球内各荧光信号的径向坐标。
- (e)每个细胞与其最近的3个邻居之间的平均距离(ncells = 128973,nspheroids = 6236)。品红色虚线指距离的第一个十分位数,青色虚线指距离的最后一个十分位数。
- (f)e部分的所有细胞核和两个划分群体对应柱状图。(品红色指第一个十分位;青色指最后一个十分位,虚线表示中值)。
- (g)细胞球的中平面共聚焦图像,红色的鬼笔环肽(phalloidin)和蓝色的DAPI染色。虚线强调了两个具有代表性的细胞。结合f和g,可以得出与细胞球核心相比,边缘附近相邻细胞核之间的平均距离更大。事实上,细胞球内部的细胞是圆形的,而在三维结构的顶部观察到一些扁平和增大的细胞,这是肝细胞球组织良好的结构特征。
- (h)归一化的ALB信号与r/R(细胞到细胞球中心的归一化距离)的关系。
- (i)h相关柱状图。从功能的角度来看,对5000个细胞球的分析表明,ALB峰的强度与细胞在三维结构中的位置相关。从r/R = 0.4
1,观察到ALB的信号强度持续增加;r/R = 00.4之间,测量到较低且平坦的信号。 - (j)一个ALB染色的细胞球的中平面共聚焦图像。白色虚线表示球体的边缘。同样显示了边缘有更强的ALB信号。
这种径向位置的ALB增加可能是琼脂糖水凝胶力学性质的结果,这与以前的观察结果一致。事实上,虽然在游离悬浮培养的细胞球中ALB的分布是相同的,但最近的研究表明,由于被胶囊刚性,被封装的肝癌细胞球的中心ALB显著减少。
通过对死亡细胞的分析,在球体边缘检测到的死亡细胞比例较高,可能是由于它们无法建立细胞与细胞之间的相互作用而被逐出细胞球。或者它们可能在细胞球形成阶段之前就已经死亡。
综上所述,该设备具有在长期静态培养下支持细胞增殖和表型保存的能力。在这项工作的以下部分,作者展示了该平台的其他三个独特功能,其中细胞测量能力可以与其他微流控方法相结合,即:
- 在控制灌注下,球体内单个细胞行为的动态监测;
- 三维和二维构型细胞的空间和时间控制共培养;
- 单个细胞球的选择性恢复。
Multiscale cytometry under dynamic drug perfusion
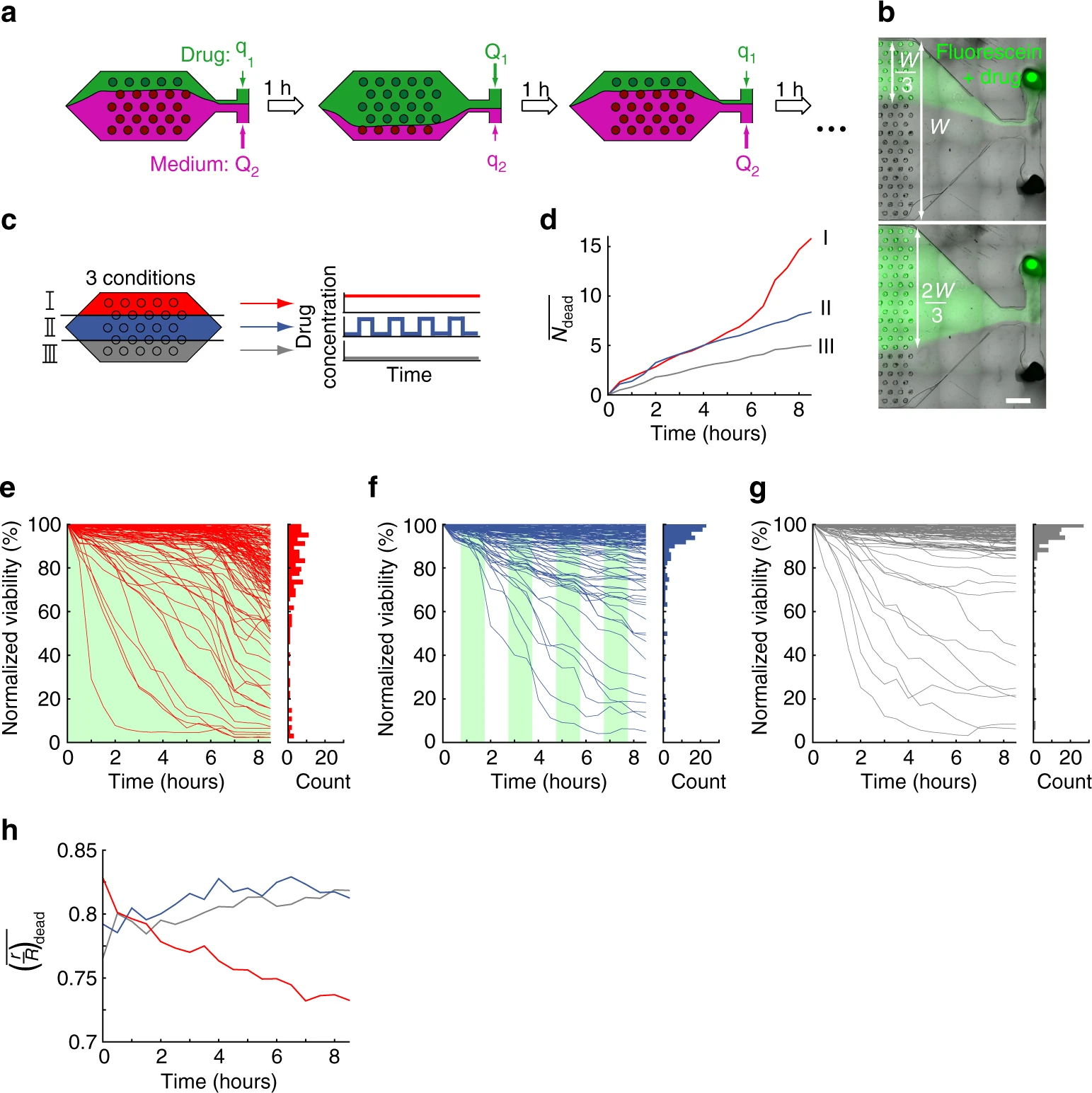
- (a)腔室内灌流两种溶液:一种只含培养基,另一种补充300 μM对乙酰氨基酚。相对流速的控制能够在空间和时间上调节细胞球对药物的暴露。
- (b)不同溶液灌注芯片的显微图。荧光素标记含有药物的溶液,W是腔室的宽度。
- (c)同一个腔室中的H4-II-EC3细胞球暴露在三种不同的培养条件下。芯片的顶部(I区)连续灌注药物(红色);中间部分(II区)间断灌注,半周期为1 h(蓝色);腔室底部(III区)灌注无药物培养基(灰色)。
- (d)平均死亡细胞数的的时间演变,由3个不同的区域中(I = 红色,II = 蓝色,III = 灰色)每个区域内的细胞球数量归一化。
- (e-g)三种实验条件下每个球体生存能力的时间演变。其中,e为区域I,f为区域II,g为区域III,绿色背景表示药物灌注。直方图显示了每种条件下实验结束时的活性分布。值得注意的是,区域III和II均有少部分细胞球在第一时间活性降低,因此均在最终表现出了大部分的高活性细胞球和少量的低活性细胞球的分布;而区域I中有部分细胞球出现了类似的快速下降情况,但药物孵育6小时后,出现了存活率普遍下降,最终活性分布图出现了更低的值及更广泛的分布。
- (h)死细胞到细胞球中心的归一化的距离的时间演变。在区域II和III,细胞死亡被观察到在细胞球的边界附近(r/R = 0.8),并在实验期间保持静止;相比之下,区域I的死亡细胞的位置开始在同一位置附近(r/R = 0.8),但逐渐向内转移,表明细胞球内部的细胞死亡率增加。
三个区域的流速恒定,并在实验期间保持在40 μL min−1。因此,流量切换不太可能导致区域II的细胞死亡。
I区细胞死亡的双重动力学表明,在持续接触药物的情况下发生两种不同的机制,一种是快速的,另一种是缓慢的。对乙酰氨基酚的细胞毒性是由N-乙酰对苯醌亚胺(NAPQI)的形成和谷胱甘肽(GSH)的逐渐耗尽引起的。H4-II-EC3产生几个参与NAPQI生产的细胞色素(Cytochrome)。其观察结果与对乙酰氨基酚毒性的已知机制一致,其发生的方式是:首先诱导肿胀样坏死(NAPQI促进的快速诱导);然后凋亡(诱导时间更长,需要消耗GSH)。在间歇区(区域II),只观察到快速机制,而没有慢速机制。这一结果表明,在区域II中,NAPQI不积累(或可能在流量切换时被冲走),而GSH保持在一个足以促使细胞存活的水平。
实验结果表明,需要连续接触对乙酰氨基酚才能诱导细胞凋亡,这可能是由于对乙酰氨基酚诱导线粒体孔开放需要时间。
Towards 3D and mixed 3D–2D organs-on-a-chip
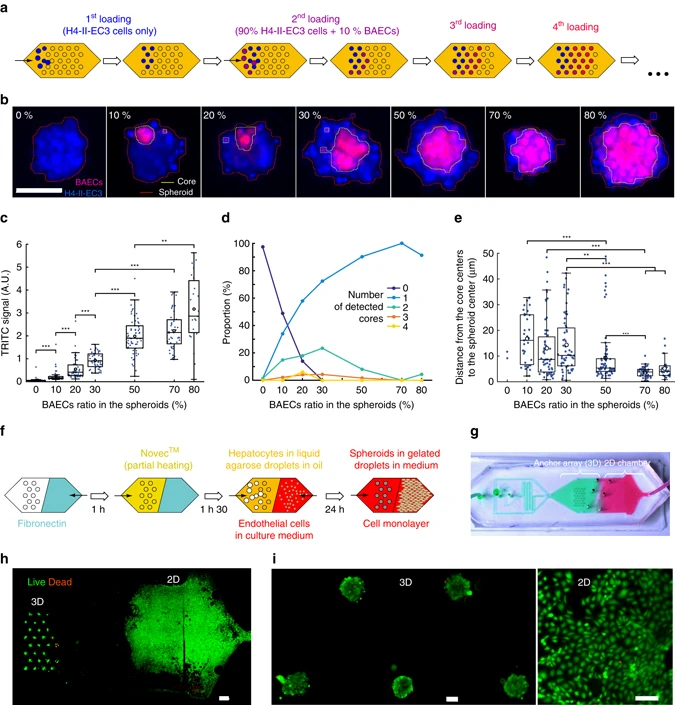
(a)在单个液滴阵列上不同条件的加载流程示意图。依次加载7种细胞溶液,其中牛主动脉内皮细胞(BAEC)的比例增加。将几种比例的BAECs和H4-II-ECs混合在一起,因此能够在同一芯片上依次形成含有不同细胞混合比例的液滴。
(b)不同BAECs比例的一个代表性细胞球的荧光图像。细胞自组装成单个球,无论细胞比例如何,BAECs都位于球的内部,被H4-II-EC3包围。
(c-e)TRITC信号(c)、细胞球中检测到的BAECs核的数量(d)和BAECs核心中心到球体中心的距离(e)与BAECs细胞比的函数关系。小比率(10-50%的BAECs)的异质肿瘤包含几个BAECs的核心,较高比率(70-80%的BAECs)则显示了一个单一的、定义明确的内皮细胞簇;高比例的BAECs形成的核心比小比例的BAECs形成的核心更可能分布在中心,后者分布在离球体中心更远的地方。
(f)3D-2D混合流程示意图:2D培养的区域涂有纤连蛋白(fibronectin),球体培养的区域涂有氟化液Novec。经过不同的表面处理后,细胞可以被装载在锚中形成细胞球或直接接种在用纤连蛋白处理的PDMS上进行2D培养。
(g)芯片设计可以实现不同的涂层溶液的空间控制,同时防止它们的混合。
(h)死活染色的肝癌细胞球和单层BAECs,显示出受控的空间分布。
(i)更高放大倍率的细胞球和二维细胞。
Selecting desired spheroids

- (a)方案示意图:用聚焦的红外激光束(橙色点)对选定的琼脂糖液滴进行局部加热。培养基溶液将细胞球运出芯片,然后将其收集在1.5 mL的试管中。
- (b)细胞球恢复的时刻图。橙色点表示激光在锚中的位置。
- (c)激光恢复后的细胞球的白光和死活染色。 在激光分选后,细胞球仍然具有活力。
- (d)在二维培养皿中重新复制(0 h)和复制后(24 h),细胞球的白光图。在二维培养皿上复制时,细胞保持着从三维结构中迁移出来的能力。
Discussion
展示了将多项技术整合到单个微流控平台,用于3D细胞培养、动态调节、多尺度细胞测量(即群体、细胞球、单细胞水平)、定量图像分析、共培养和选择性恢复。从几秒钟到几天都可以观察到相同的细胞,不需要对流程进行任何修改,任何细胞球都可以在任何时候被提取。
Reference
Sart S, Tomasi R F-X, Amselem G, et al. Multiscale cytometry and regulation of 3D cell cultures on a chip: 1[J]. Nature Communications, 2017, 8(1): 469.