一种体外生成可灌注微血管网络的稳健方法。
Introduction
思路:
- 工程微血管网络(Engineered microvascular networks,MVNs)已成为体外组织和器官功能研究中普遍的工具。
- 体外形成的MVNs的价值在于它们能够概括体内微血管生理学的主要功能,并阐明关键生理和病理过程的机制,例如选择性渗透性、血管发生和血管新生、血脑屏障、高血糖效应等。
- MVNs必须是可灌注的,才能在体内复制类似的功能。血管内流动由两侧培养基通道提供,但微柱之间的血管必须在培养基通道界面处打开。
- 然而,有利于在微柱之间形成开放血管的条件通常会导致MVNs的直径大于典型的微血管。目前已经发表的文献中,可灌注的MVNs既有毛细血管样的血管,也有比其大得多的血管。
- 为了可靠地形成具有所需毛细血管样几何形状的可灌注的MVNs,作者调查了接种参数对MVNs几何形状的影响,在此过程中,建立一个强大的两步接种策略,能够为整个中心区域的微柱间开放血管和毛细血管样MVNs生成提供有利条件。
Vasculogenesis是指血管发生(有人译为血管生成),尤特别指胚胎早期由中胚层来源的成血管母细胞(angioblasts)在间充质(mesenchyme)内,直接分化为血管始原细胞(endothelial precursors),再空腔化形成血管的过程。这过程是从无到有的“全新(de novo)”形成过程,重点在直接分化、从无到有;
Angiogenesis一般常译为血管新生,它是由原来的血管经由出芽(sprouting)方式,分支出新的血管。这過程由VEGF(血管内皮生长因子,vascular endothelial growth factor)导引,不管是胚胎期或生命期,新组织的形成和生长都伴随此种血管新生成,来扩大原有血液供应网络。像许多肿瘤都会利用血管新生方式,来增加局部组织供血量,以满足癌细胞不断分裂过程中时时需要的营养供给。
可以这样想,血管发生(vasculogenesis)是由0变1,而血管新生紧跟之后,由1+1+….。血管新生是一种添加性生长,而血管发生则是全新、从无到有的分化发育。
来自知乎:MeiZhou晨星的回答。
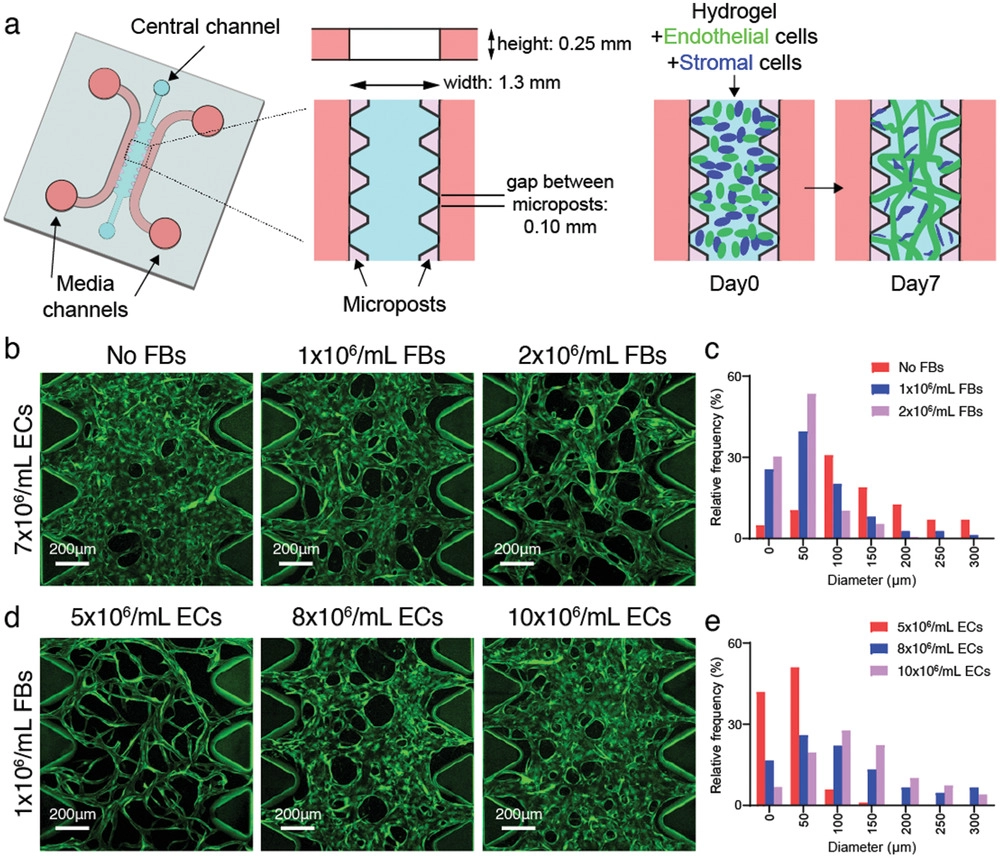
- (a)用于MVNs形成的带有微柱的微流控装置的示意图。该装置通过一排紧密间隔的微柱来划分相邻的通道,微柱上的表面张力使注入的液体保持在其通道内,因此内皮细胞(ECs)和基质细胞的悬浮液在中央通道中保持分隔。几天之内,ECs通过自组装过程生成相互连接的类似毛细血管的MVNs。
- (b)显示永生化人脐静脉内皮细胞(ImHUVEC,7×106 mL-1)MVNs的代表性图像,这些MVNs分别与0、1或2×106 mL-1浓度的肺成纤维细胞(Lung FBs)共培养。其中,ImHUVECs表达蓝色荧光蛋白(BFP)。
- (c)不同Lung FBs浓度共培养的ImHUVEC MVNs的微血管直径分布。与没有基质细胞支持和相互作用形成的MVNs相比,与基质细胞(如成纤维细胞FBs)共同接种的ECs产生的MVNs直径更小,长期稳定性更好。
- (d-e)不同浓度表达BFP的ImHUVECs产生的MVNs的代表性图像(d)和直径分布(e)。较低浓度的ECs使血管直径分布更接近生理范围(<10 μm)。
Initial Seeding Parameters Control MVN Geometry In Vitro
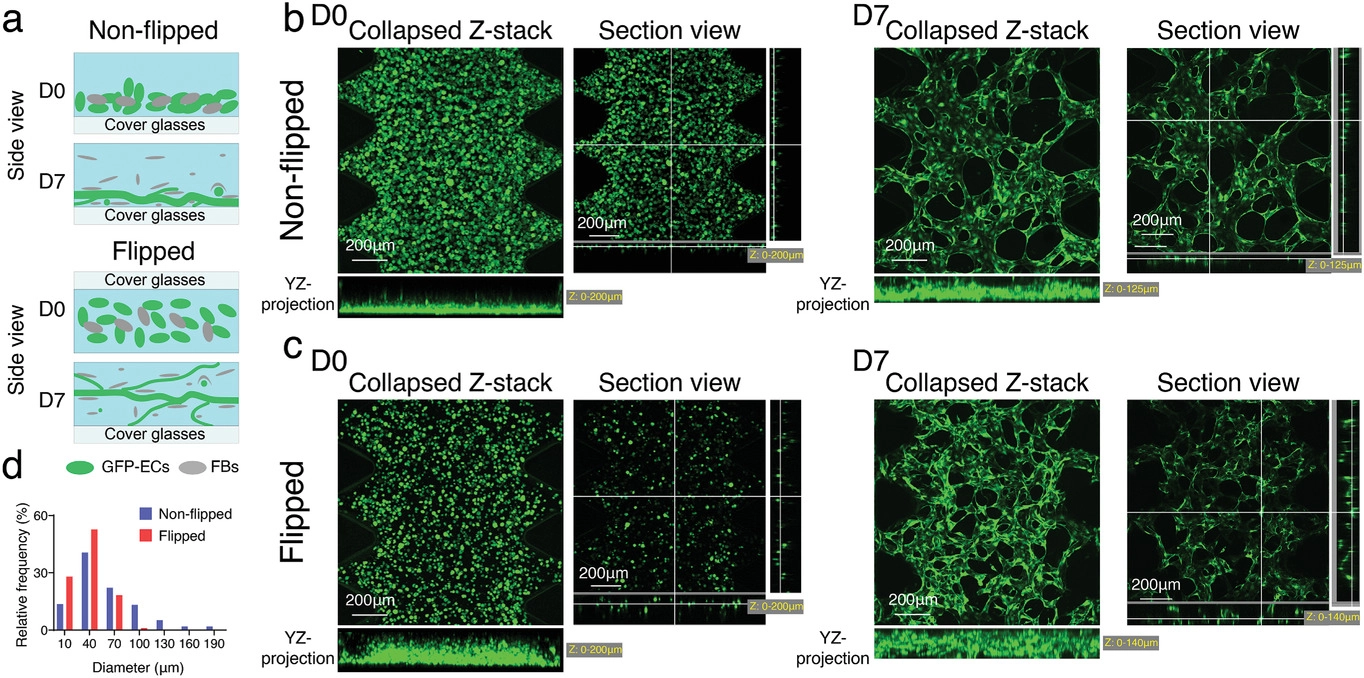
- (a)非翻转和翻转接种方法的示意图。在水凝胶固化过程中,细胞由于重力作用下沉到装置底部,这导致在装置底部附近产生较大直径的扁平MVNs。通过在水凝胶聚合过程中多次(3~5次)翻转设备来抵消ECs的汇集,可以改善细胞分布,并产生更3D分布的MVNs,其直径更接近体内毛细血管的直径。
- (b-c)在第0天(左)和第7天(右),在非翻转(b)或翻转(c)方法中MVNs的代表性共聚焦图像。
- (d)以非翻转或翻转方法产生的MVNs的直径分布。与非翻转方法相比,翻转方法使细胞混合物在所有三个维度上分布更均匀,从而在第7天产生更窄和更多的3D分布式MVNs。
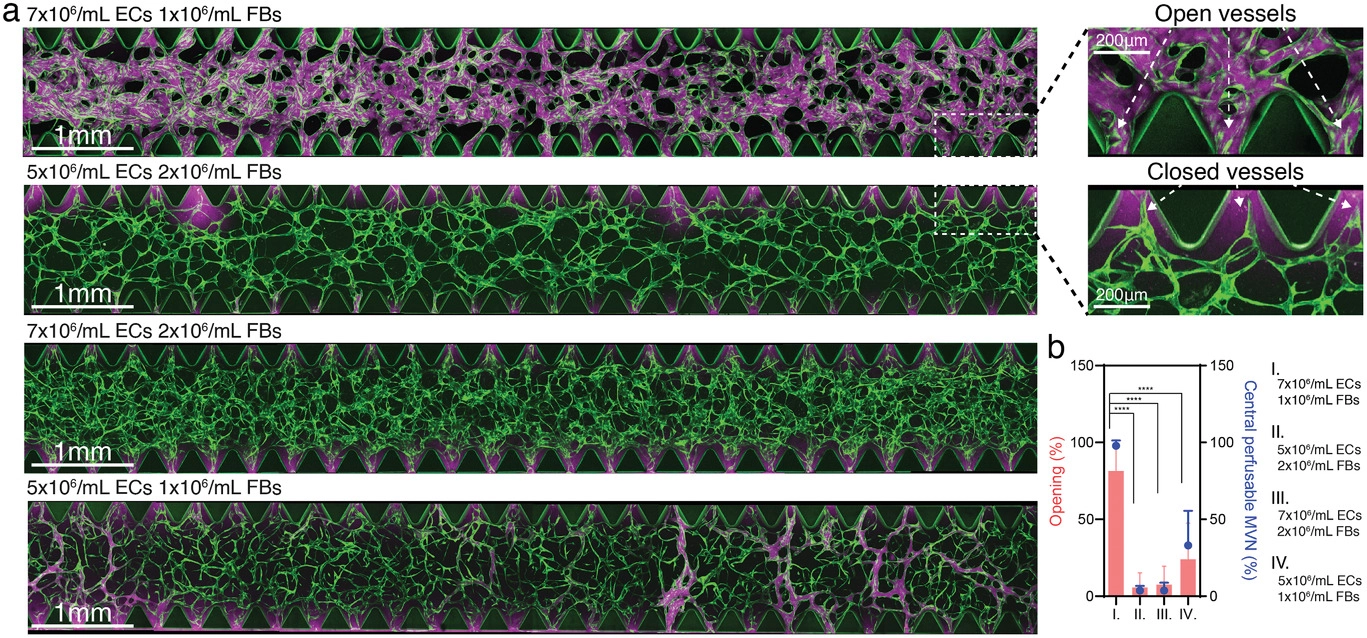
- (a)由不同初始浓度Lung FBs/ImHUVECs形成的MVNs的代表性共聚焦图像(左)。微柱之间区域的开放和闭合微血管的放大视图(右)。较低浓度的ECs和较高浓度的Lung FBs将导致更多不可灌注的MVNs形成(即较大直径的MVNs更具备可灌筑性)。
- (b)各种EC和Lung FB浓度下的MVNs的微血管开放百分比(柱)和中央可灌注MVNs百分比(点)的统计分析。
选择葡聚糖(70 kDa)是因为它的分子量接近人血浆白蛋白(分子量约66.5 kDa),是人血浆中的主要蛋白质,适合用于测试内皮屏障功能。
Two-Step Method to Generate Perfusable MVNs with Physiological Geometry

- (a)带有微柱的微流控装置中的两步接种方法示意图。所有细胞都悬浮在水凝胶前体(纤维蛋白原fibrinogen/凝血酶thrombin)中。首先,将高密度的EC接入装置中。随后吸出未固化的凝胶,由于表面张力留在微柱之间区域的ECs称为外层ECs。然后,将EC/Lung FB接种到中央凝胶区域。多次翻转微流控装置以改善中心的血管细胞3D分布。MVNs将在7天内形成,中心区域的EC自组织成直径较小的MVNs,而较高浓度的外层EC形成更宽、更开放的管腔。
(b)形成MVNs的传统一步法的示意图和代表性图像。
- 非翻转、较高的EC(8×106 mL-1)和较低的Lung FB(1×106 mL-1)密度倾向于形成可灌注但直径更宽的MVNs。
- 翻转、较低的EC(5×106 mL-1)和较高的Lung FB(1.5×106 mL-1)密度形成不可灌注、直径较窄的 MVN。
(c)生成中间直径较窄的可灌注MVNs的两步法的示意图和代表性图像。在本实验中,使用BFP-ImHUVECs(青色,10×106 mL-1)作为外层ECs;将带有Lung FB(1.5×106 mL-1)的GFP-ImHUVECs(绿色,5×106 mL-1)接种在中间。白色箭头指向狭窄的微血管。
- (d)由一步或两步法产生的MVNs的直径分布。
- (e) 通过一步或两步法产生的MVNs的开口百分比(柱)和中央可灌注 MVN 百分比(点)的统计分析。
Applications of MVNs Made with Two-Step Method
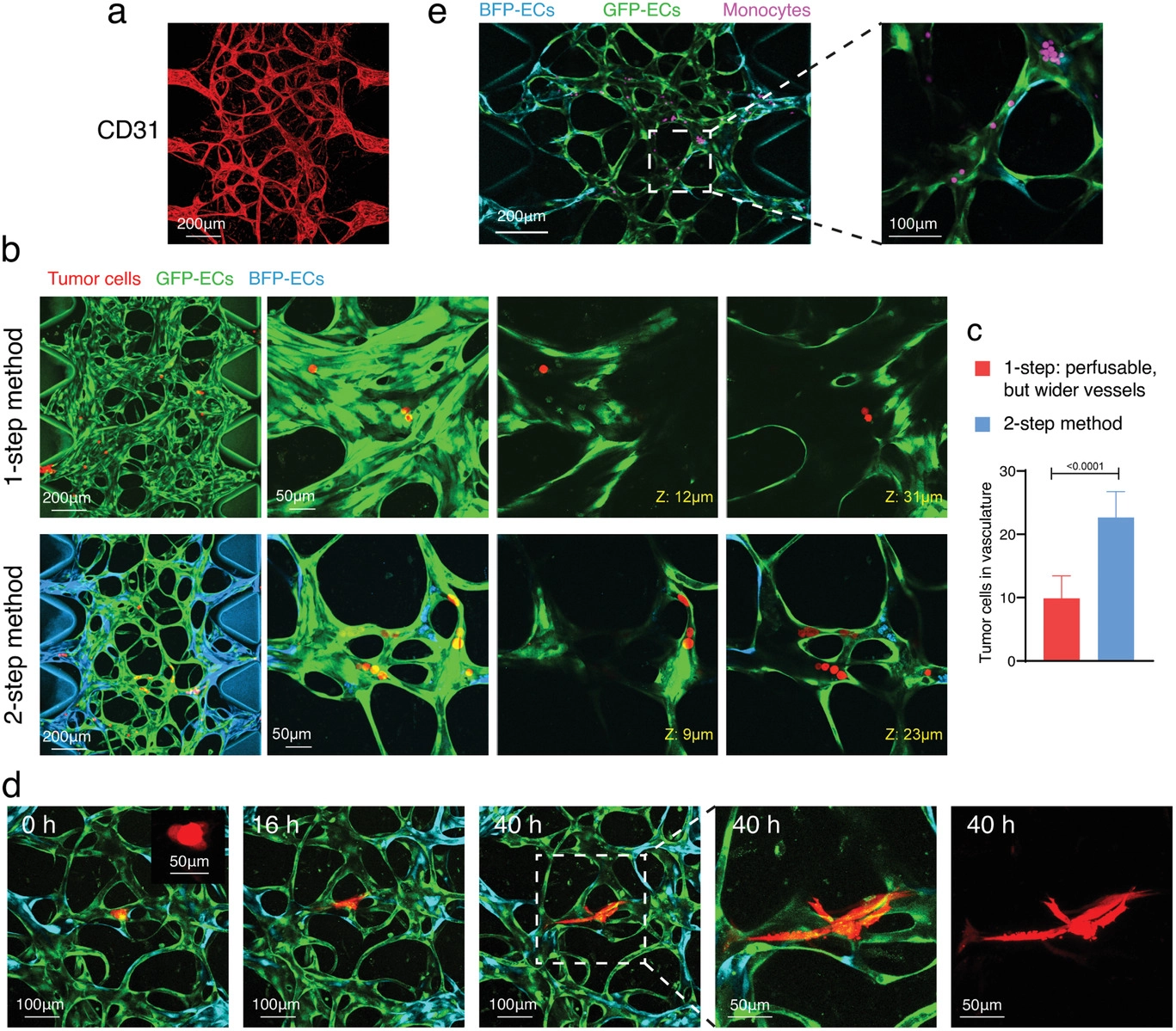
- (a)用两步法产生的ImHUVECs/Lung FBs MVNs用CD31抗体染色,以证明该MVNs适用于免疫荧光染色。
- (b)MVNs中灌注肿瘤细胞(红色)的代表性共聚焦图像,分别用一步或两步法产生ImHUVECs/Lung FBs MVNs。右侧的放大视图显示了在这两个MVNs中的H69M肿瘤细胞。
- (c)通过一步或两步法产生的MVNs中的肿瘤细胞数量量化。显示更多的肿瘤细胞通过两步法形成的MVNs。
- (d)由H69M细胞系制成的肿瘤簇(2-4 个细胞,模拟循环肿瘤簇)在两步MVN中灌注并成像40小时。
- (e)单核细胞在两步法产生的ImHUVECs/Lung FBs MVNs中进行灌注,作为免疫细胞灌注研究的一个例子。
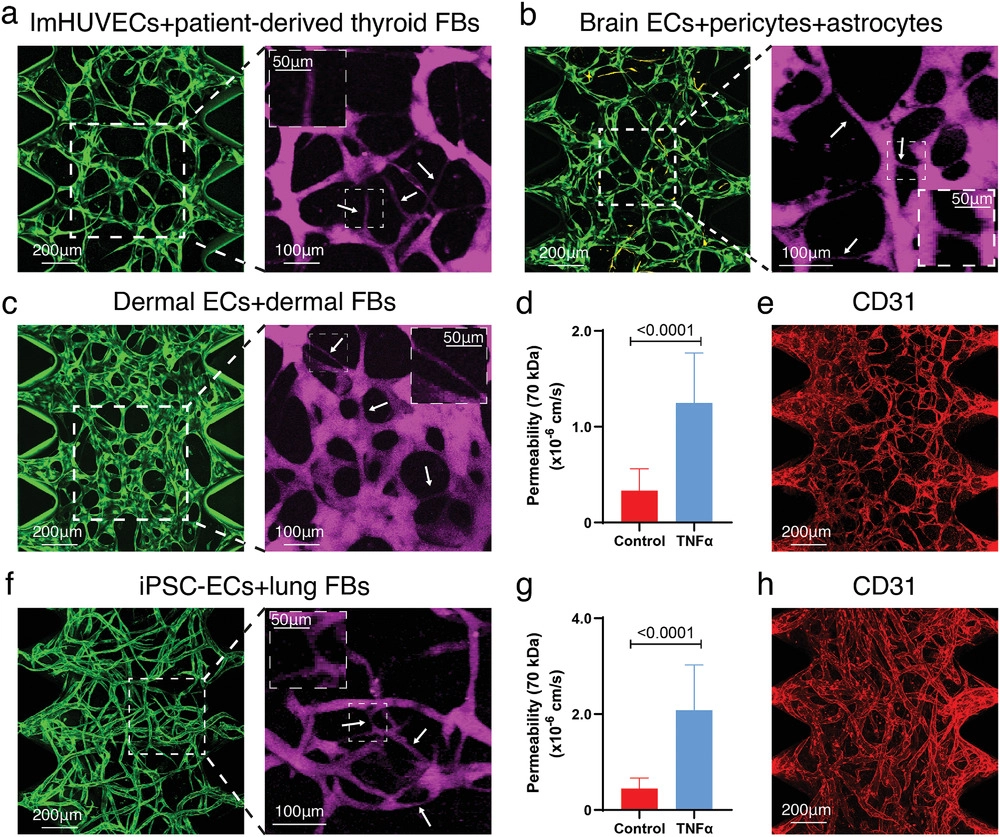
- (a)ImHUVECs与患者衍生的甲状腺成纤维细胞(thyroid FBs)共培养形成的两步法MVNs。
- (b)脑ECs与脑周细胞和星形胶质细胞形成的两步法脑MVNs。其中活化星形胶质细胞的的免疫荧光染色呈黄色,10 kDa葡聚糖用于脑MVNs灌注。
- (c)人真皮血液微血管ECs(HDBMECs)和真皮FBs形成的两步法真皮MVNs。
- (d)肿瘤坏死因子α(TNF-α)处理与否的两步法真皮MVNs的渗透性分析。TNF-α能作用于内皮细胞,导致其作用紊乱,渗透性升高。
- (e)用CD31抗体(红色)染色的两步法真皮MVNs。
- (f)由iPSC衍生的ECs和Lung FBs形成的两步法MVNs。
- (g)TNF-α处理与否的两步法iPSC-ECs MVNs渗透性的统计分析。
- (h)用iPSC-ECs和Lung FBs制成的两步法MVNs的CD31免疫染色(红色)。
Hybrid MVN Formation using Two-Step Method
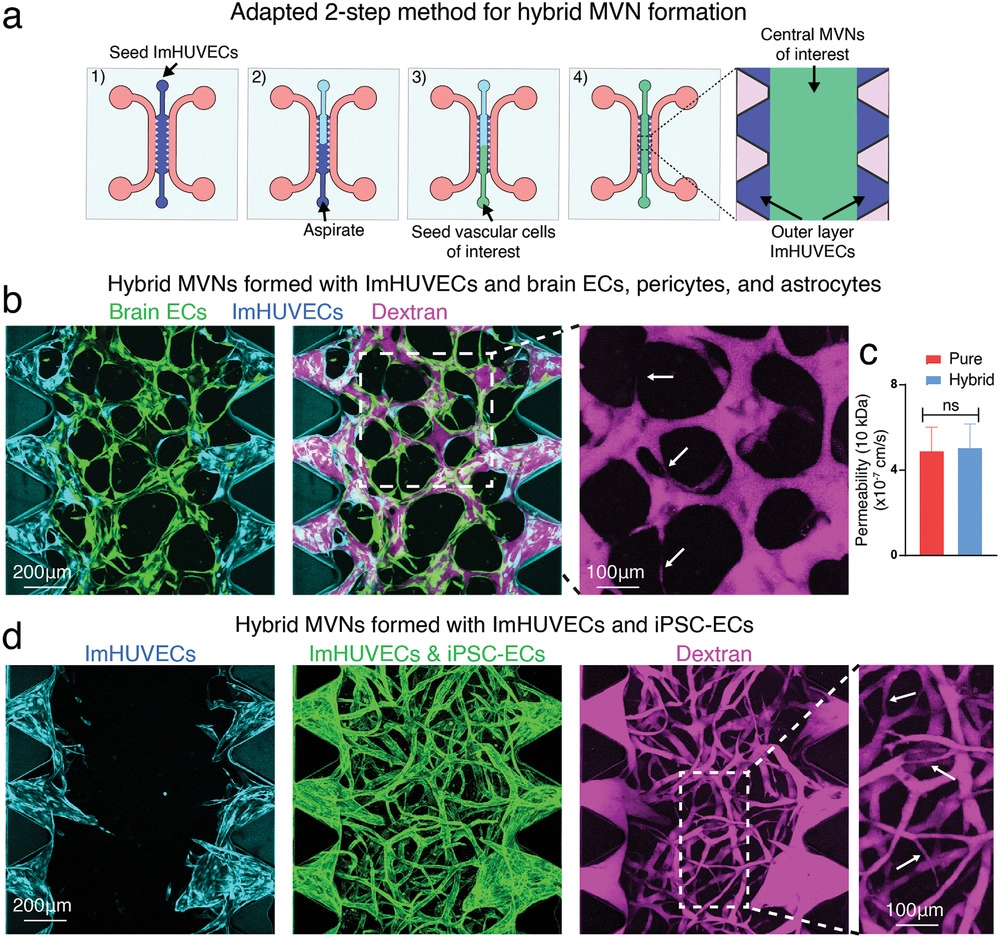
- (a)微流控装置中采用的两步播种法示意图。为了节约使用可能难以大量获得的对MVNs形成感兴趣的血管细胞的使用,ImHUVECs用于外层ECs播种,在吸出未固化的ImHUVECs/凝胶前体后,将感兴趣的血管细胞(如脑原代ECs以及星形胶质细胞和脑周细胞)接种到中央凝胶区域。
- (b)由ImHUVECs(外层)和大脑特异性MVNs(中间区域)形成的混合MVNs的代表性图像。
- (c)使用10 kDa葡聚糖通过两步法产生的单一或混合脑MVNs中心区域的渗透率测量。
- (d)由ImHUVECs(外层)和iPSC-ECs/Lung FBs(中间区域)形成的混合MVNs的代表性图像。
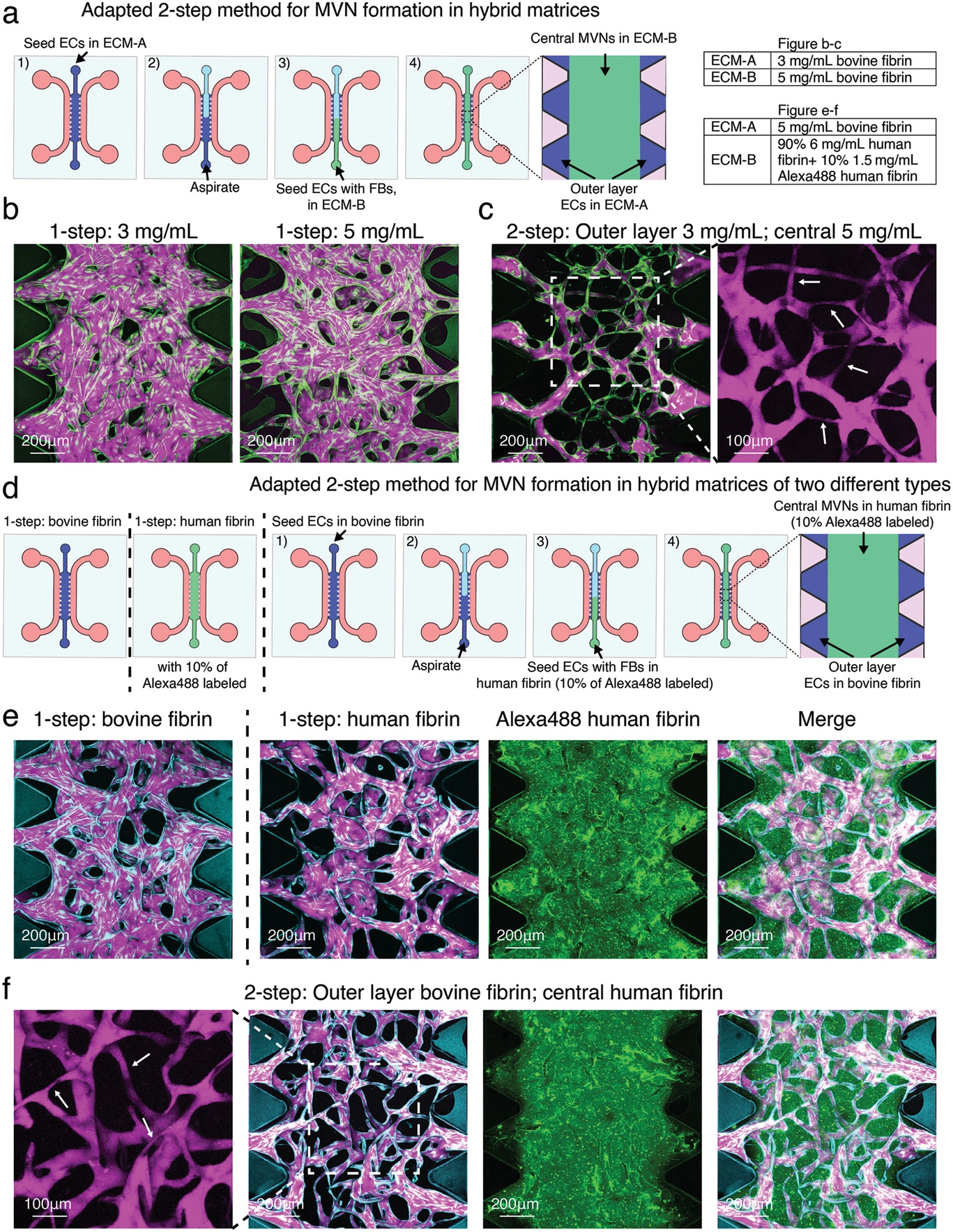
- (a)适用于两种不同类型ECM(ECM-A和ECM-B)的两步接种法示意图。为了减少稀缺ECM(如患者来源或组织特异性ECM)的使用,常见的ECM(如牛纤维蛋白bovine fibrin)可用于外层EC播种。在吸出未固化的ECs/ECM-A液体后,将含有ECs/FBs的稀有ECM-B接种到中央凝胶区域以在混合ECMs中形成MVNs。
- (b)使用一步法在3 mg mL-1 ECM-A或 5 mg mL-1 ECM-B的纤维蛋白中形成的宽的可灌注MVNs的代表性图像。
- (c)使用两步法(外层3 mg mL-1 ECM-A,中央区域5 mg mL-1 ECM-B)形成的MVNs的代表性图像。
- (d)在牛或人纤维蛋白中接种MVNs的实验设计。为了区分两种类型的ECM,用Alexa Fluor 488标记人纤维蛋白。
- (e)使用一步法形成的MVNs的代表性图像。
- (f)使用两步法形成的混合MVNs的代表性图像。
Discussion
- 包括免疫细胞在内的血细胞会通过直径小至5–10 µm的毛细血管。循环肿瘤细胞和簇在转移过程中也会通过机械捕获和内皮粘附在毛细血管中。因此,具有毛细血管样几何形状和尺寸的可灌注MVNs对于体外研究正常和病理过程至关重要。
- 基于微流体的毛细血管网络形成方法已成为研究单个毛细血管水平生理学的普遍工具。 但已有研究未能构造出更接近毛细血管直径的MVNs,其原因在于较大直径的MVNs才能具有可灌注可能性。
- 作者开发了一个简单的两步法,可以增加MVNs向培养基通道开放的可能性,同时保持毛细血管样形态。
- 作者发现ECs与FBs浓度的比率是血管尺寸的主要决定因素之一,较低的值有利于更窄、更生理的微血管。
- 作者发现大约7天FBs往往出现在微柱之间的区域,这表明FBs从中心区域迁移到外层区域。
- 作者发现由各种类型的血管细胞形成的两步法MVNs显示出不同的几何形状,这种差异强调了为特定研究建立组织特异性MVNs的重要性。
- 使用两步法形成MVNs的一个缺点是浪费了形成外ECs层的细胞,可以在外层ECs中使用永生化的HUVECs或其他容易获得的ECs。
- 两步法也可以适用于在混合ECM中形成MVNs。已经表明,肿瘤内的ECM密度和硬度高于正常组织,但密度更高的ECM会产生更窄的微血管,因此可以通过在外层和中间区域使用不同的ECM密度来潜在地解决这个问题。
Conclusion
作者开发了一种两步的、用于形成具有生理相关网络几何形状的功能性和可灌注的MVNs新方法。
Reference
Wan Z, Zhong A X, Zhang S, et al. A Robust Method for Perfusable Microvascular Network Formation In Vitro[J]. Small Methods, 2022.