一种利用3D生物打印构建神经母细胞瘤(Neuroblastoma,NB)的体外血管模型,用于探索内皮化微环境中的肿瘤功能。
Introduction
思路:
神经母细胞瘤是儿童期最常见的颅外实体瘤,由发育中的交感神经系统引起,约占所有儿科肿瘤死亡的15%。多年来,对NB肿瘤微环境(TME)的重要性及其对治疗反应和肿瘤行为的影响的研究越来越多。
尽管NB的体内小鼠异种移植模型对于药物测试和验证是最有效的,但缺乏有效的体外或离体平台来概括NB TME一直是高效和实时动态研究的障碍。
3D打印和生物打印在设计各种TME模拟物方面受到越来越多的关注。
- 其优势在于可以对生物材料进行稳健地空间控制,从而能够创建高度复杂和异质的3D结构,模拟经常发生的高度血管化的肿瘤组织;
- 其缺点是打印方法的制造过程相对缓慢和复杂,以及可以模拟TME内的细胞外基质(ECM)的可用生物墨水数量仍然有限。
目前的生物打印癌症模型仍然面临着几个重大挑战,包括:
- 由于水凝胶生物墨水固有的物理和生物学特性,结构保真度和再现性低;
- 印刷细胞密度低,远低于生理肿瘤环境;
- 只能打印相对简单的组织结构,很少概括复杂、动态和血管化的TME。
作为球体培养的NB细胞已被证明可以更好地模拟体内实体瘤的生长特征。最近,保留细胞-细胞和细胞-环境相互作用的3D多细胞肿瘤球已逐渐用于概括自然3D TME。通过将EC与癌细胞混合,3D混合癌症球表现出癌症的一些主要标志,包括癌症侵袭和血管生成。这种策略的主要缺点是其有限的生理相关性,仅简单地混合内皮细胞和癌细胞而没有表现出TME的分层模式。
该研究使用嵌入式3D生物打印技术设计和制造了具有多个血管通道的水凝胶立方模型,具有高结构保真度和可重复性的复杂3D结构。该NB-EC模型在静态和动态条件下培养,评估了与肿瘤发生相关的变化,包括细胞活力和生长、肿瘤大小和形态以及 NB-EC整合。
Schematic illustration of the research workflow in this study
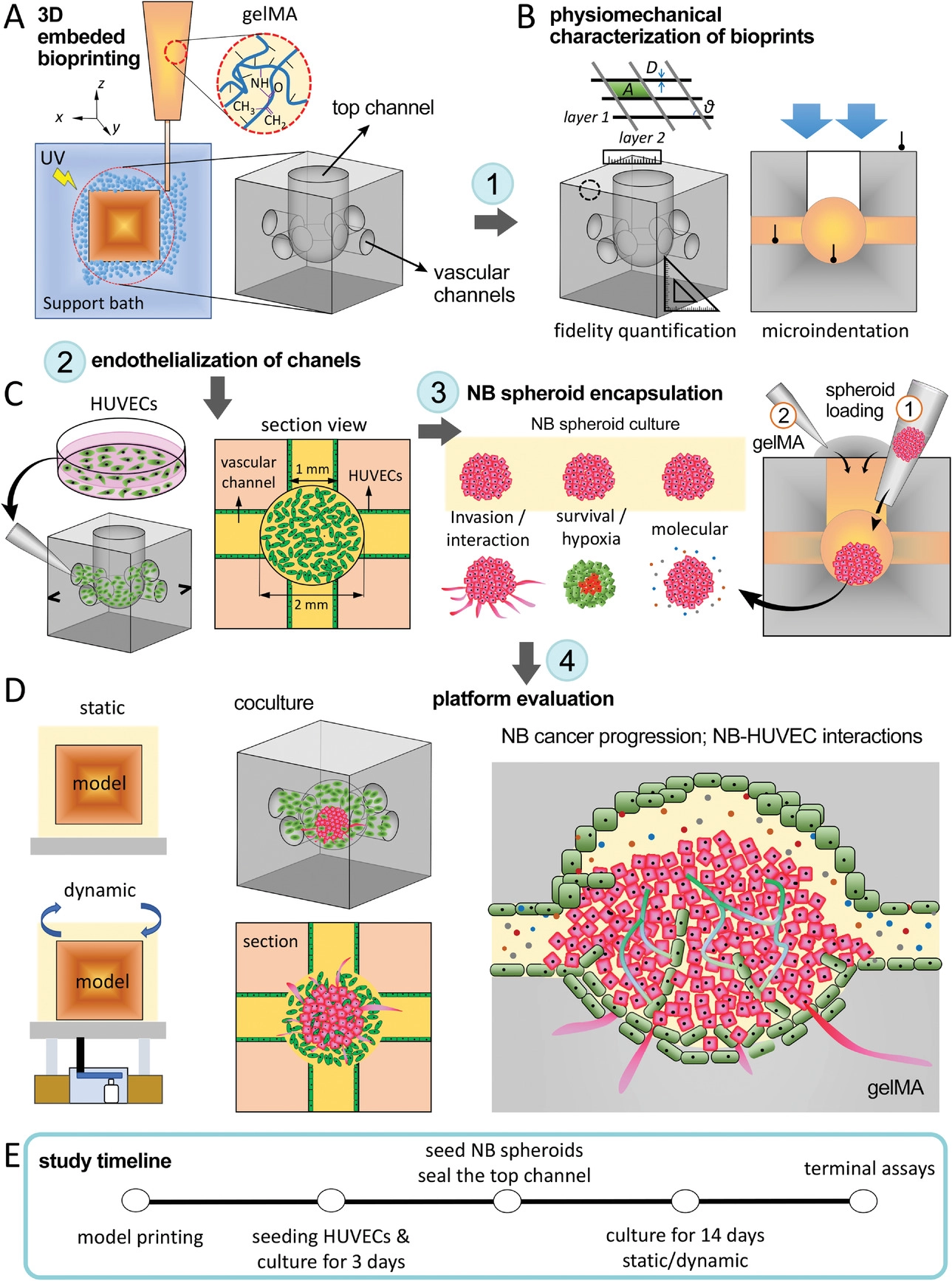
- (A)甲基丙烯酸酰化明胶(gelMA)组织模型的 3D 嵌入式生物打印,由侧面的互连通道网络(用作脉管系统)和顶部的一个通道(用于形成NB球)组成。
- (B)生物打印的gelMA结构的物理力学表征。通过测量几个结构参数来量化打印保真度,包括线径 (D)、线间表面积( A)、线均匀度(U)和层间角度 (α),由CAD文件中每个参数的基线值标准化。通过无限制压缩以及显微压痕测定来测量gelMA结构的机械性能。
- (C)将HUVECs(左)和NB球(右)接种到3D生物打印结构中。人工将HUVECs接种到侧通道的管腔表面上以形成血管内皮。再通过顶部通道将NB球结合到每个内皮化gelMA构造的中心腔中(步骤1),最后通过交联的gelMA铸造和密封顶部通道腔(步骤2)。
- (D)在静态与动态(摇摆)培养条件下评估NB-HUVEC平台。在与HUVECs相互作用时观察了NB肿瘤的进展。
- (E)本研究中使用的关键实验步骤的时间表。
Characterization of printing fidelity and mechanical properties of bioprinted gelMA constructs

- (A-B)生物打印的gelMA结构的打印保真度和机械性能的表征。设计(A)和生物打印(B)用于评估2D保真度。
- (C)2D平面模型的打印保真度表征。
- (D)使用优化的打印条件,打印了大量的立方血管gelMA模型。
- (E-F)3D打印gelMA的打印保真度表征。
- (G) 在不同的紫外线照射时间下,从不同位置测量gelMA模型的弹性模量。30 s和 1 min的紫外线照射,接近天然NB组织的刚度,由于这两个条件刚度没有显著差异,选择具有更好地可打印性的1 min曝光作为条件。
NB spheroid survival, growth, and interaction with the 3D matrix in the bioprinted gelMA construct
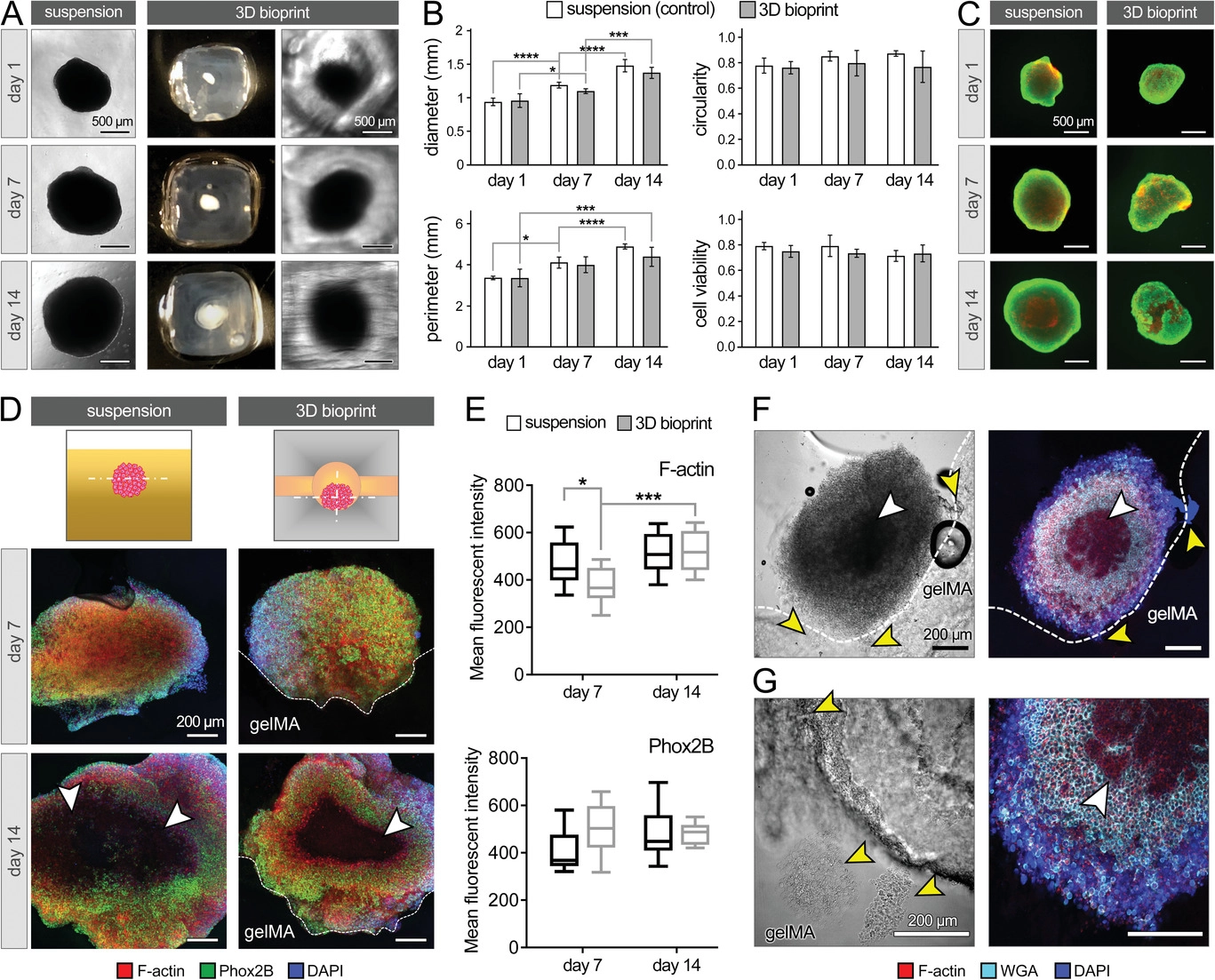
- (A)NB球从第1 d到第14 d在悬浮(对照)与3D gelMA组中的生长。
- (B)NB球直径、周长、圆度及活性的量化。
- (C)对悬浮的整个NB球(左)与3D生物打印(右)进行活/死染色。结果证明了生物打印通道结构在2周的体外培养中保持NB球活力和生长。
- (D)在第7 d和第14 d对培养的NB球进行免疫组织化学分析,染色F-肌动蛋白(红色)、Phox2B(绿色)和DAPI(蓝色)。白色箭头突出显示球体中心的坏死核心。
- (E)基于(D) 中的共聚焦图像,得到的F-肌动蛋白(顶部)和Phox2B(底部)的定量平均荧光强度。发现在第7 d,gelMA 模型中的F-肌动蛋白表达低于对照组,但在第14 d显著增加至可比较的水平,而对照组随着时间的推移表现出稳定的表达。表明3D gelMA封装的初始效果,阻碍了F-肌动蛋白的形成,从而阻碍了NB细胞在前7 d的迁移和扩张(侵袭),但在培养的第二周有所缓解。
- (F-G)NB细胞和gelMA在培养第14 d的相互作用,通过染色F-肌动蛋白(红色)、WGA(青色)和DAPI(蓝色)。白色箭头突出显示球体中心的坏死核心。黄色箭头指向NB细胞侵入到gelMA基质中。结果证实了生物打印的通道结构能够为NB肿瘤生长提供兼容和支持的微生态位,以及评估有助于侵袭的细胞特性的模型。
F-肌动蛋白 :与细胞迁移高度相关;
PHOX2B:NB分化和干性维持的关键介质;
WGA:标记糖蛋白,用于活细胞或固定细胞的质膜成像。
Evaluating HUVECs viability and growth within the 3D bioprinted gelMA channels
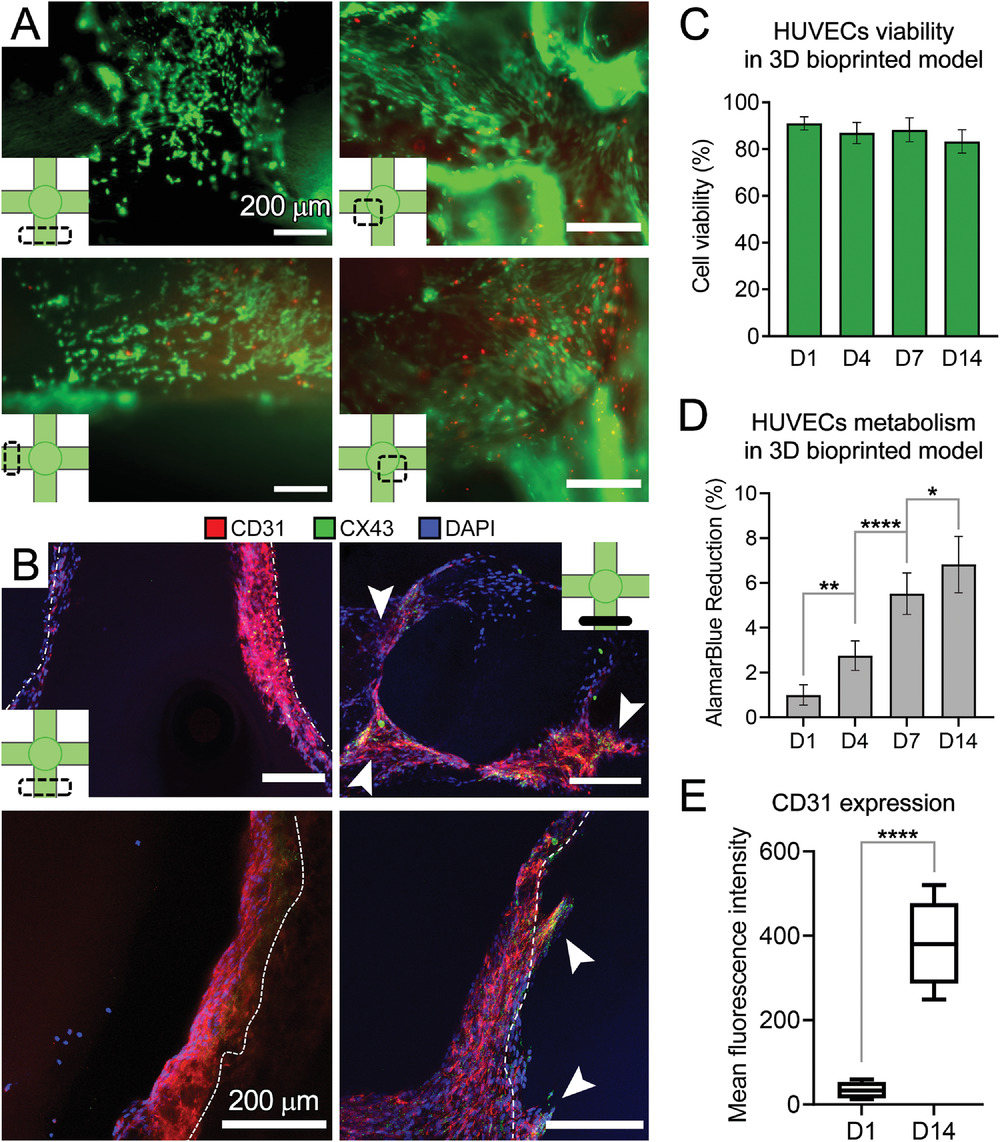
- (A)培养第14 d 3D通道结构内各个区域的活/死染色结果表明通道内皮化。
- (B)从纵向(左)和切向/垂直(右)视图对HUVECs进行CD31(红色)、缝隙连接蛋白43(connexin-43、CX43、绿色)和DAPI(蓝色)染色。可见HUVECs从通道的管腔表面渗入到gelMA基质中(白色箭头),进一步证实了gelMA的生物活性以支持EC功能。
- (C)在培养的第1、4、7和 14 d定量活/死检测。
- (D)用于测量HUVECs代谢活性的AlamarBlue测定,作为细胞活力和生长的测量值,结果表明生物打印中HUVECs的活力/增殖增加。
- (E)在(B)中的共聚焦图像上进行的CD31荧光强度的量化。表明在gelMA生物打印中ECs的持续生长和增强的细胞功能,可能是高度多孔和相对柔软的支架结构为ECs提供了一个合适的微环境来附着、迁移、重塑和分泌它们自己的ECM。
AlamarBlue:即阿尔玛蓝,细胞增殖过程中,细胞体内的环境由氧化环境变化成还原环境,呼吸链中的NADPH/NADP, FADH/FAD, FMNH/FMN和NADH/NAD的比值升高。alamarBlue®的活性成分resazurin (刃天青)是一种无毒、可透膜的蓝色染料,有微弱的荧光性,它作为一种氧化还原指示剂,被细胞内吞后,在细胞质中被以上这些代谢中间体还原,其还原产物resorufin呈现出粉红色并有很强的荧光性,最后可用普通分光光度计或荧光光度计进行检测,吸光度和荧光强度与活性细胞数成正比,因而可作为细胞增殖和细胞毒性定量检测的一个理想的指示器。
NB-HUVEC coculture in 3D bioprinted gelMA constructs
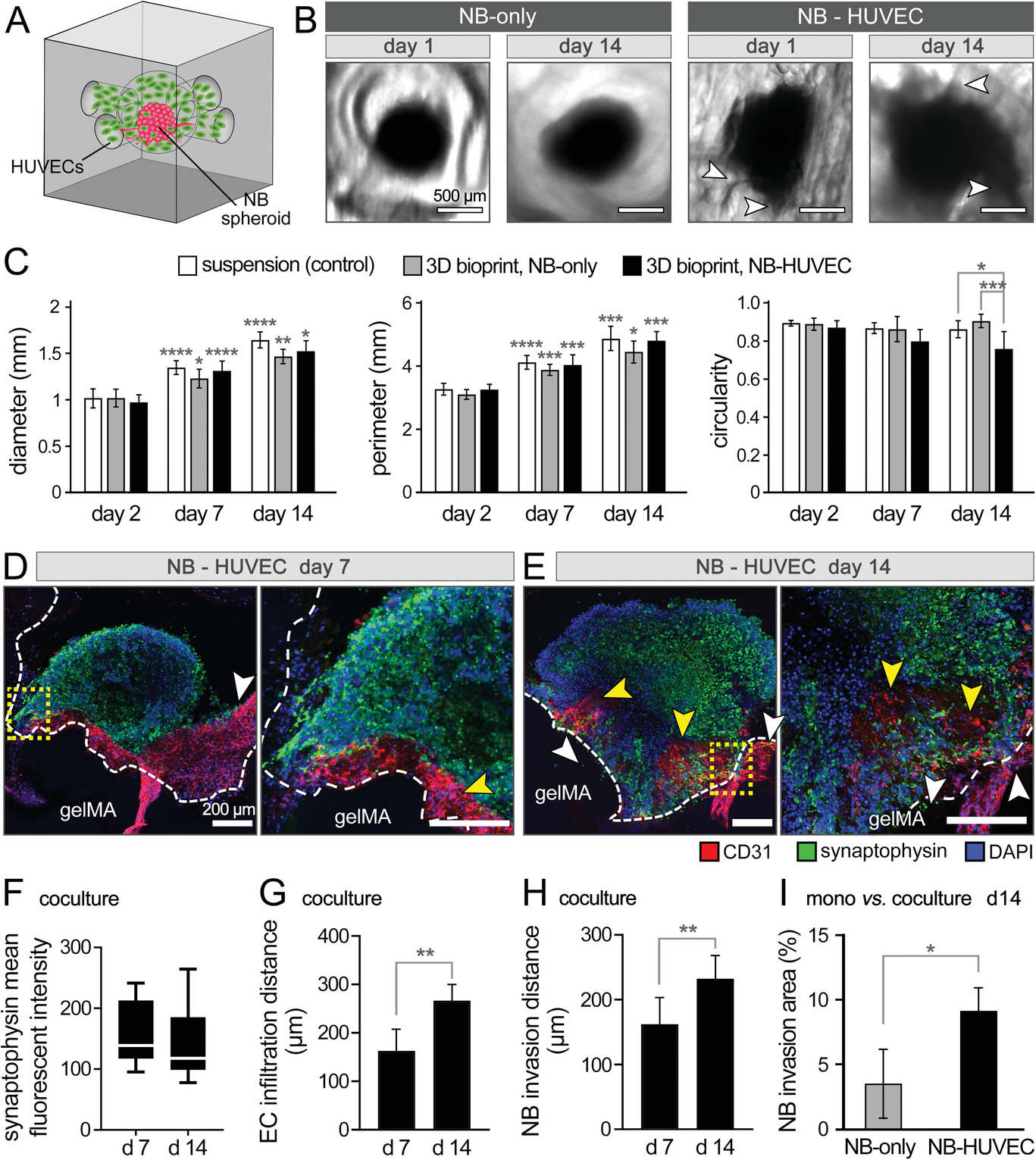
- (A)NB-HUVEC在3D生物打印gelMA结构中的共培养示意图。
- (B)在不存在(仅NB)和存在HUVECs(NB-HUVEC)的情况下,在gelMA结构中培养1 d和14 d的NB球的白光图像。
- (C)NB球形状的量化,即悬浮培养的球体的球体直径、周长和圆形度(对照)与那些在有或没有HUVECs的生物打印模型中生长的球体比较。与其他两组相比,共培养球的圆度显着降低,可以得出结论,添加ECs不会阻止NB生长,但可能会将球的形状改变为不对称形式。
- (D-E)在第7 d(D)和第14 d(E)对共培养球进行免疫组织化学成像。共聚焦图像显示了EC特异性标志物CD31(红色)、突触素(synaptophysin、绿色)和DAPI(蓝色)的染色结果。白色箭头指向NB侵入 gelMA,黄色箭头突出EC渗透到球中,gelMA边界由白色虚线描绘。结果表明EC可能促进了NB细胞的入侵。
- (F-H)基于共聚焦图像的突触素(F)、NB细胞侵入到gelMA(G)中的距离和EC到癌球体(H)中的浸润距离的量化。其中,共培养的NB在NB特异性突触素表达方面没有显着差异,表明EC没有诱导不良反应。
- (I)在培养第14 d对单培养(NB-only)与共培养(NB-HUVEC)中NB细胞侵入gelMA基质的程度进行量化,报告为球外gelMA组织的%面积。
NB spheroids cocultured with HUVECs in 3D bioprinted models under static and dynamic conditions
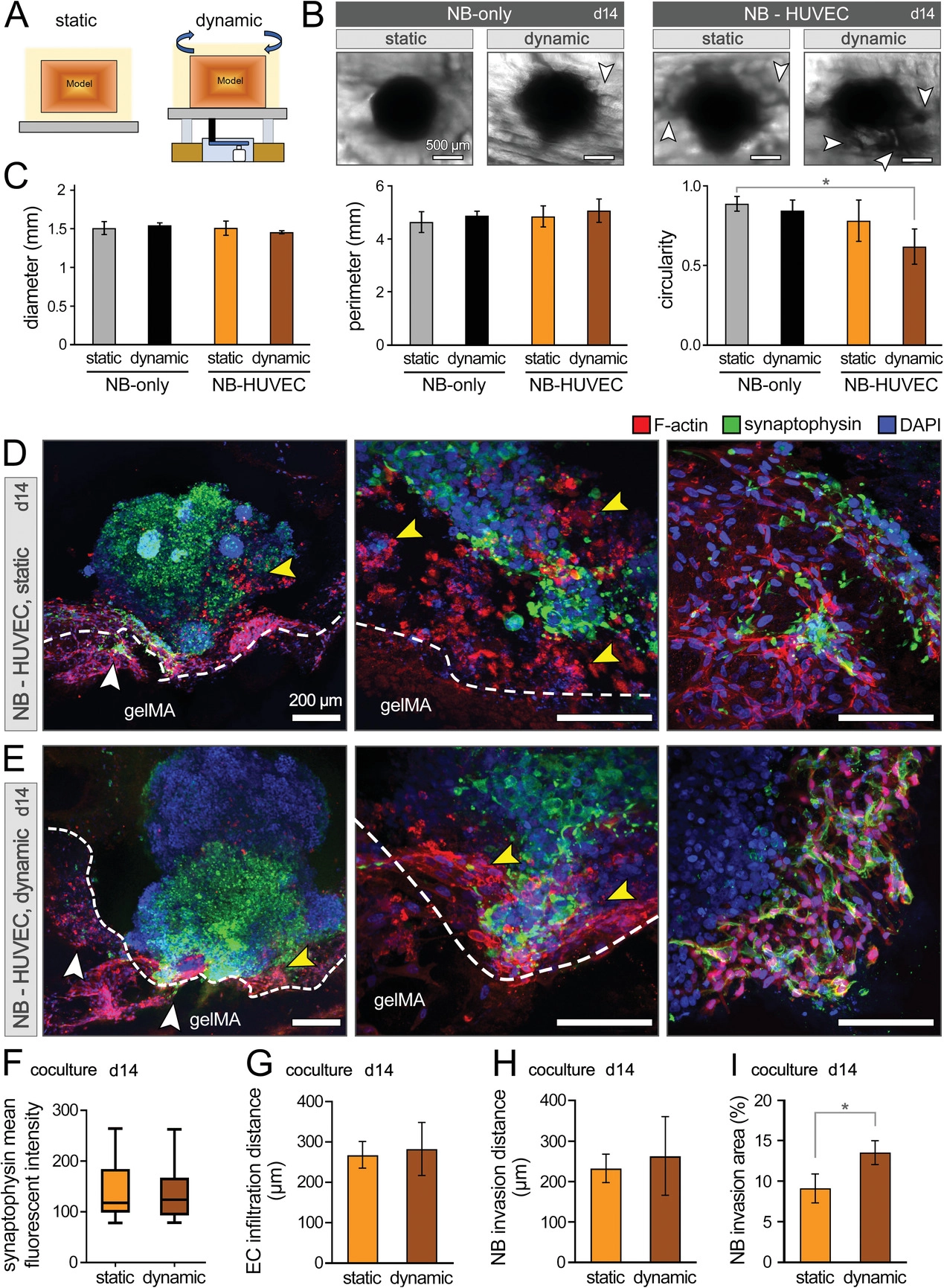
- (A)在静态和动态条件下,在3D生物打印模型中与HUVEC-NB的实验装置方案。
- (B)在静态和动态培养14 d后,仅NB和NB-HUVEC共培养组中NB球形态的白光图像。白色箭头突出显示NB细胞对gelMA基质的侵袭。
- (C)NB球形状在直径、周长和圆度方面的量化。动态培养会导致共培养的HUVEC-NB圆度进一步降低。
- (D-E)第14 d在静态(D)和动态(E)条件下对共培养模型进行免疫组织化学分析。不同放大倍率的共聚焦图像显示了NB-EC-gelMA基质相互作用,包括NB细胞对gelMA的侵袭(白色箭头)和EC渗入NB球(黄色箭头)。
- (F-I)在共培养的第14 d,NB细胞突触素表达的量化(F)、EC浸润到NB球中的距离(G)、NB细胞浸润到gelMA基质中的距离(H)和以gelMA的面积百分比表示的NB细胞浸润程度(I)。其中NB细胞侵袭程度显着增加。
Analysis of metabolic, angiogenesis, and gene expression profiles of 3D bioprinted in vitro NB models
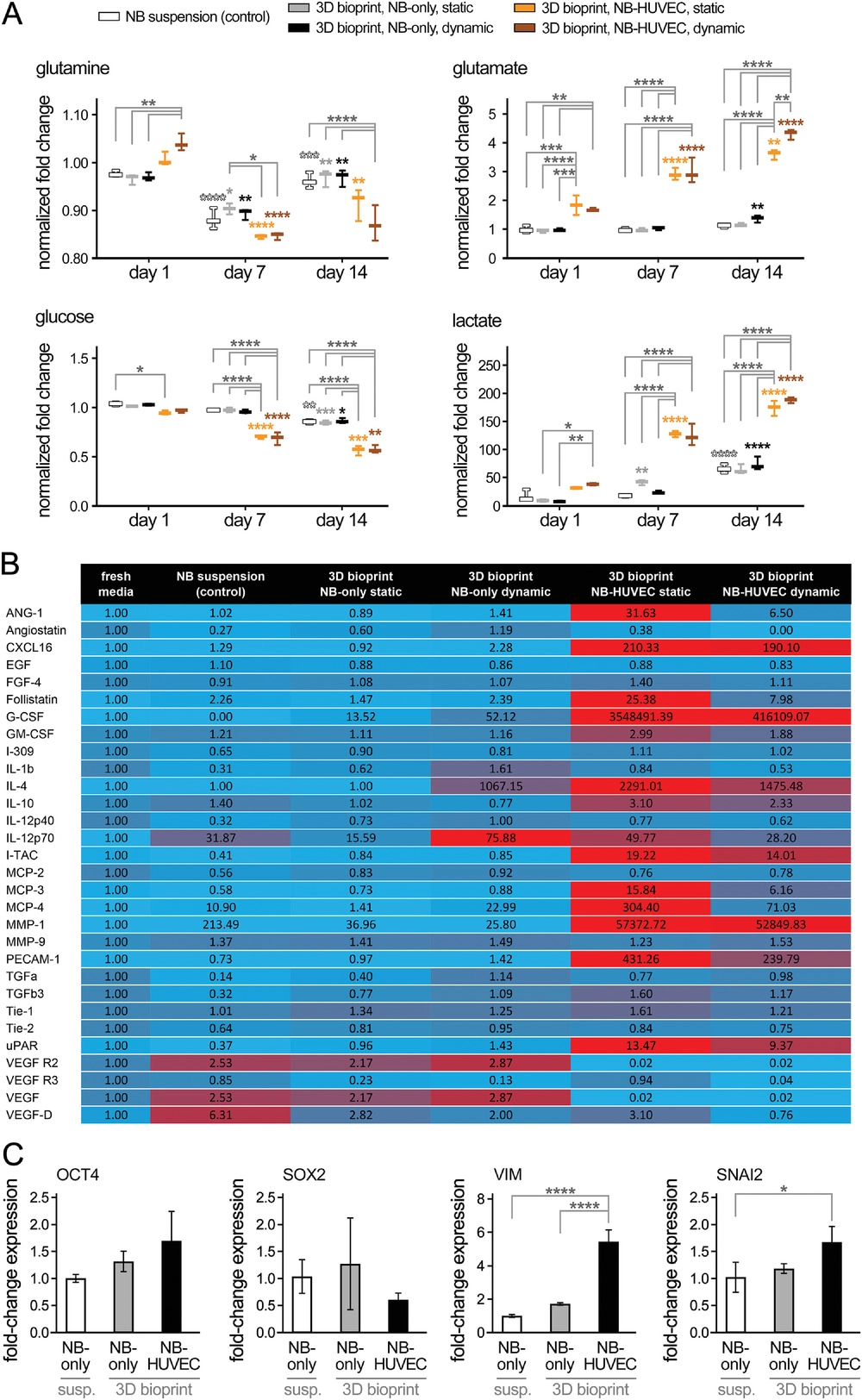
- (A)分析3D生物打印体外NB模型的代谢、血管生成和基因表达谱。在培养的第1、7和14 d收集的上清液(培养基)上进行营养物质(谷氨酰胺glutamine和葡萄糖glucose)消耗和代谢物(谷氨酸glutamate和乳酸lactate)积累的纵向生物分析测定。与仅NB组(悬浮对照和立方体中的球体)相比,NB-HUVEC在静态和动态培养中均表现出显著更高水平的营养代谢物消耗量和伴随代谢物的产生和积累。共培养组的新陈代谢增加可归因于细胞总数的增加(添加ECs),以及掺入的ECs的更高活性(生长)。与静态培养相比,动态共培养组的养分消耗和废物产生水平略高,这在第14 d谷氨酸产生的情况下变得显著,这可归因于在灌注(动态)与静态培养的3D生物打印肿瘤模型中促进了代谢物的交换。
- (B)在培养第14 d进行的各组的血管生成分析(热图)。收集培养物上清液,与偶联抗体一起孵育,并使用微阵列扫描仪进行分析。这些结果表明3D生物打印gelMA模型中的静态NB-HUVEC培养物表现出最高水平的血管生成活性。与动态共培养相比,静态共培养的更高的血管生成和增殖谱可归因于减少/扰动的流动和剪切应力在增加血管氧化应激中的作用,因此增殖和血管生成的进展。
- (C)通过qRT-PCR分析对每个条件下分离的NB球进行相对基因表达(OCT4、SOX2、VIM和SNAI2) 分析。结果表明,与干性或间充质特性有关的基因,例如OCT4和SOX2,在所有组中都是稳定的,这表明在所有条件下都保持干性特性。其他对上皮-间质转化至关重要的基因,例如VIM和SNAI2,在共培养条件下显著增加,支持血管化生物打印模型中NB更具间充质或侵袭性表型。
Summary, Conclusions, and Future Works
- 癌细胞和血管内皮之间的相互作用对于重塑TME和癌症转移是必不可少的。为了在体外概括这些过程,先进的3D 生物打印技术与3D细胞培养技术相结合,在建立具有高结构准确性和可重复性的平台方面显示出巨大的希望。
- 本研究介绍了一种先进的生物打印方法,能够生物制造具有高结构保真度和可重复性的血管肿瘤模型,为直接掺入NB球、内皮细胞和其他TME组件提供高度可调的微环境。
- 结果证明了生物打印血管模型在动态培养条件下维持NB肿瘤活力、生长、侵袭和与内皮相互作用的可行性。在gelMA结构中培养的NB-HUVECs表现出增强的细胞间相互作用,NB细胞向gelMA组织迁移/侵袭,EC向肿瘤缺氧区域浸润。代谢、细胞因子和基因表达分析表明,动态条件下的 NB-EC 共培养模型最有效地反映了肿瘤细胞和脉管系统之间的相互作用。
- 未来的研究可以利用这个生物工程平台来充分表征NB(和其他)肿瘤的生物学特性,以响应TME参数的时空变化,例如免疫和基质细胞、缺氧、血管生成及ECM刚度和组成。
Reference
Ning L, Shim J, Tomov M L, et al. A 3D Bioprinted in vitro Model of Neuroblastoma Recapitulates Dynamic Tumor-Endothelial Cell Interactions Contributing to Solid Tumor Aggressive Behavior[J]. Advanced Science, 2022, 2200244.