一种基于微流控的方法,用于在存在定义的免疫细胞群的情况下对肿瘤球体的命运进行多重分析。
Introduction
思路:
细胞毒性T淋巴细胞(cytotoxic T lymphocytes,CTL)消除肿瘤细胞的能力是开发肿瘤免疫疗法的基础,例如免疫检查点抑制剂(抗CTLA-4、抗PD1或抗PD-L1单克隆抗体)和细胞疗法(CAR T细胞)的发展。多种参数会影响患者对免疫疗法的反应,但CTL的数量、表型和分布,可能在几种类型的癌症中具有很强的预测价值。
几个关键问题仍未得到解答:
- CTL如何遇到肿瘤细胞并在肿瘤微环境(TME)中积累?
- CTL杀伤的动力学是什么?
- 肿瘤根除需要多少CTL密度?
- 单个CTL是在TME中自主行动还是相互作用?
了解决定肿瘤块是否消退的基本原理对于设计、优化和评估肿瘤免疫治疗策略至关重要。
有多种方法可用于评估T细胞对肿瘤的细胞毒性:
- 基于细胞悬液中的体外测定已用于测量CTL杀伤能力,并且在单细胞水平上进行时,可提供有关T细胞群内功能异质性程度的信息。然而,这些体外检测缺乏3D肿瘤微环境的复杂性,而后者强烈影响T细胞的行为和功能。
- 活体成像提供了对发展中肿瘤内单个T细胞的动力学、信号传导和杀伤行为的直接洞察。然而,这些方法的局限性包括它们提供了在有限的空间和时间窗口中的交互视图。事实上,连续观察期通常仅限于几个小时,无法全面了解TME中的T细胞历史。
先进的体外模型可以概括TME的某些方面,同时提供了对系统动力学的访问,如类器官和器官芯片。但从这些系统中收集可以解释CTL攻击结果的一般规则仍然具有挑战性,因为它需要将T细胞行为的定量测量(其本质上是随机的)与肿瘤细胞命运联系起来。
在这里介绍了一种基于微流控的方法,用于在存在定义的免疫细胞群的情况下对肿瘤球体的命运进行多重分析。该方法基于对静止微流体液滴内数百个肿瘤球体的平行形成、操作和观察。当与数学模型相关联时,时空解析数据的数量和质量使研究者能够查明导致球体破坏的关键行为,并检测和了解肿瘤结果的异质性。
Parallelized immune challenge on an integrated microfluidic chip
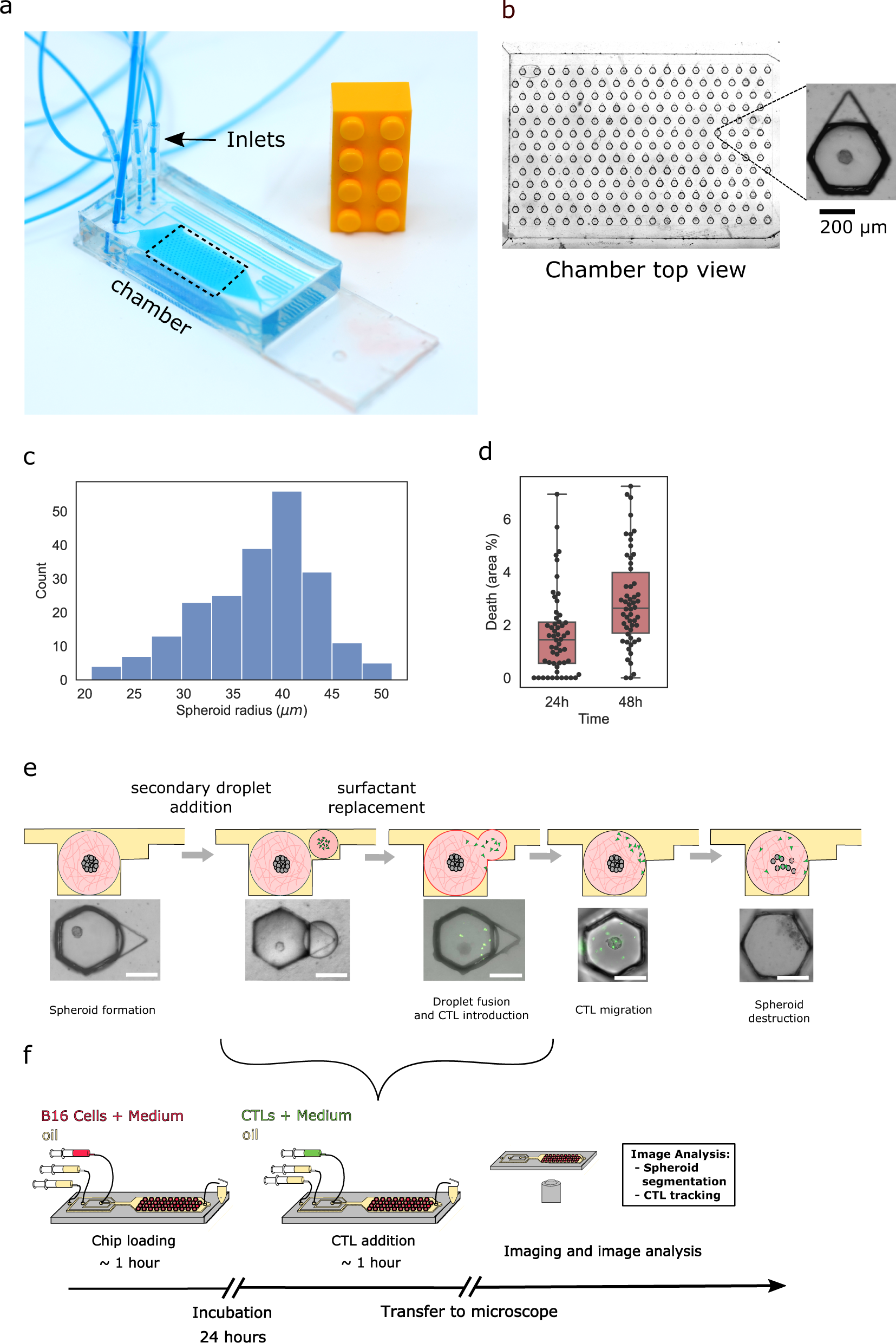
- (a)标准载玻片上的微流控芯片。
- (b)芯片捕获区域的放大图(虚线框)显示了234个捕获液滴的阵列。每个液滴在Matrigel中包含一个表达卵清蛋白的小鼠B16黑色素瘤(B16 OVA)球,如插图所示。
- (c)单个芯片内的球体半径分布。大多数情况下获得的球体半径在35~45 μm之间。
- (d)在24和48 h后使用活死染色进行活力测量。活死染色显示,在芯片中48 h后,不到3%的细胞死亡。
- (e)示意图显示了一个带有肿瘤球的初级液滴,然后添加并融合了一个含有GFP标记的CTL的次级液滴,最终导致肿瘤细胞死亡和球体破碎。
- (f)完整实验方案的示意图。
CTL migration in micro-device reproduces in vivo behavior

- (a)左图为具有Matrigel液滴内瞬时速度矢量的CTL的代表性图像。右图为一个液滴中超过24 h的CTL轨迹,每种颜色代表一个单独的细胞轨迹。黑色虚线圆圈勾勒出球体边界。
- (b)三个不同T细胞的代表性速度随时间的变化图。记录的速度显示出运动和停滞阶段的交替周期。
- (c)在固定时间步长Δt = 1 min期间,细胞迁移距离(Δr)的概率分布。
- (d)具有和没有球体的CTL迁移的均方位移(MSD)。发现这均没有显示出显著差异,因此这两种情况下CTL都经历了超扩散随机游走。
- (e)显示最初CTL与球体靠近和接触的时间序列。
- (f)单个CTL在基质胶和球体表面迁移时的轨迹。颜色图表示细胞的瞬时速度。
- (g-h)在凝胶和球体上迁移的细胞的(g)平均速度和(h)均方位移指数(α)。显示出CTL与肿瘤球接触后发生了行为变化。
- (i)在基质胶和球体上迁移的细胞的均方位移。粗体和虚线表示球体和基质胶中CTL的MSD的最佳拟合,指数分别为1.1和1.4。
A positive feedback loop drives CTL accumulation on the spheroid

- (a)实验中CTL与球体第一次接触时间的分布和随机迁移的CTL的理论分布。
- (b-c)在每个球体上检测到的(b)CTL数量和(c)比例作为时间的函数。每条细线代表一个跟踪的球体,粗体是平均值。红色是B16 OVA球体的积累,蓝色是B16 WT球体。
- (d)两个代表性球体上CTL的数量与时间的函数关系,显示检测到附着/脱离事件。
- (e)随机累积模型示意图:CTL可以以不同的概率从凝胶切换到球体。pin(Δt) (conv. pout(Δt)) 是细胞在时间间隔Δt内附着到(conv. 脱离)球体的概率。计算实验中的附着和脱离事件使我们能够推断出速率λin和λout。
- (f)B16 WT(蓝色)和B16 OVA(红色)细胞的附着率(λin)、脱离率(λout)和亲和力比(λin/λout)。
- (g)B16 WT和B16 Ova球体的亲和力比与球体上检测到的CTL数量的函数。
- (h)归一化的附着率λin(白色)和脱离率λout(红色)与附着在球体上的CTL数量的函数。B16-OVA球体上存在的CTL数量越多,比率越高,因此,积累速度越快,这是正反馈的标志。
Killing of B16 cells by CTLs is heterogeneous

- (a) 显示球体上CTL(OT1)位置、Caspase 3/7荧光死亡标记和B16细胞碎片的代表性序列。3 h处的白色箭头表示CTL旁边出现了Capsase信号。在9 h它代表一个碎片化的死细胞。
- (b)显示给定球体与CTL相互作用的关键事件的时间表:球体上CTL的接触时间、Caspase 3/7信号的检测、碎片事件的检测。
- (c)第一个Capsase信号的时间与第一次观察到的细胞破碎拟合。结果表明这两个观察结果密切相关,因此,用白光图像来量化球体杀伤,从而简化分析流程。
- (d)定义14 h内出现第一个碎片事件为“成功杀死”,WT(黑色)和OVA(红色)球体在14 h内显示至少一个碎片事件的百分比。对成功杀死的统计影响的分析表明,主要参数是液滴中的CTL数量和到达球体的CTL数量。
OT1:见知乎,指能特异性结合OVA的CD8细胞。
Tumor spheroid killing involves collective effects

- (a)两张代表性图像,显示第一次碎片事件期间球体上的CTL簇。白色圆圈在破碎细胞周围的半径为30 μm。通过观察显示有碎片的球体上的CTL积累速率更快。
- (b)碎片区域周围半径≤30 μm内的CTL数量分布。平均发生细胞破碎的CTL数约为3.4,当只有1个CTL时,几乎不发生细胞破碎。
- (c)总结观察到的趋势的草图:CTL(绿色)在球体表面迁移并在特定区域聚集在一起,观察到B16细胞(红色)的碎裂。
- (d)在时间间隔Δt内,连接有n个CTL的球体具有破碎的概率ΓfragΔt。
- (e)说明球体上CTL数量和碎片出现的可能情景。
- (f)碎片率Γfrag可以建模为独立CTL与球体相互作用的结果,每个CTL的单个碎片率值为ρ,因此得到独立CTL模型的概率。相反,可以将碎片率视为协作过程的结果。
- (g)Γfrag的估计值作为球体n上CTL数量的函数。与独立CTL模型(蓝色虚线)的结果相比,实验测量(黑点)符合指数(红色虚线)吻合,表明CTL与靶细胞之间独立相互作用的模型不符合球体破碎的潜在机制,即存在协同效应。
Long and short-range interactions combine to determine probability of CTLs to kill a tumor spheroid

- (a)浓度依赖性杀伤被建模为两种互补机制的结果:CTL对目标位点的长距离协同吸引和球体上的局部杀伤合作。
- (b)液滴中球体命运的演变可以建模为一个分支过程:在每个时间步长,球体都可能以ΓfragΔt的概率破碎,并且CTL可以附着或脱离球体。因此作者首先根据实验得出的参数λin和λout在每个时间点模拟球体上CTL数量的演变,再基于CTL的数量和Γfrag计算球体破碎可能,对每个时间间隔重复该过程,迭代更新球体上的T细胞数量和球体状态(碎片或完整),直到模拟实验的时间已经失效。
- (c)使用(b)中的模型和上面获得的参数对实验的第一次碎片时间与模拟的时间进行比较。阴影区域代表模拟数据的95%置信区间。粗线代表平均值。在这两种情况下,随着液滴中CTL数量的增加,第一次碎裂时间会减少,并且模拟值与实验观察到的碎裂时间非常吻合。
- (d)实验(黑点)和模拟(蓝线)球体碎裂概率作为液滴中CTL数量的函数。阴影区域代表模拟数据的95%置信区间。粗线代表平均值。模拟不仅证实了CTL数量在导致球体破碎中的关键作用,而且模拟和实验测量之间的密切吻合表明球体破碎过程很好地概括了这两种机制:球体上CTL的集体积累和局部协同杀伤。
Discussion
现有的用于癌症-免疫相互作用的微流体模型将每个芯片视为一个单独的实验并使用传统的生物测量技术研究。本研究引入了一种从体外实验中提取生物信息的新范式,将并行实现视为“蒙特卡洛实验”,以概率方式达到不同的结果。
液滴提供了几个独特的功能:
- 在明确定义的时间合并许多液滴对的能力,从而提供了并行实验的共同开始时间。
- 液滴内的封装可以很好地控制每个平行实验中的条件,从而允许在单个设备上进行大规模多路复用实验。
这些技术优势与概率建模相关联,通过将CTL的时空动态与肿瘤球体的结果相关联,推断有关CTL感知和响应肿瘤的能力的关键生物学信息。作者发现随机发生的第一次CTL与肿瘤细胞接触会触发一个正反馈回路,导致CTL在球体上加速积累。然后CTL在球体上形成簇,增强了它们杀死靶细胞的能力,导致肿瘤破碎。
几种机制可能解释了协作CTL积累和杀死:
- 由T细胞感知和产生的趋化因子能够驱动它们的蜂群行为,其中多个亚致死性细胞毒性协同诱导靶细胞杀伤。
- 初始CTL-肿瘤细胞相互作用可通过肿瘤微环境中的IFN-γ产生和扩散增加MHC class I表达来促进肿瘤破坏。
作者的方法有助于支持和产生新的假设,随后可以在分子水平上进行剖析。
Reference
Ronteix G, Jain S, Angely C, et al. High resolution microfluidic assay and probabilistic modeling reveal cooperation between T cells in tumor killing: 1[J]. Nature Communications, 2022, 13(1): 3111.