一种协同尺寸排阻和局部准静态动力学的双层微孔芯片,用于超高通量的单细胞RNA测序。
Introduction
思路:
单细胞RNA测序(scRNA-seq)在解开复杂细胞生态系统中的异质转录谱方面取得了巨大进展。
目前已有的单细胞分离策略,例如:
- 手动分离法:耗时耗力,通量低;
- C1仪器:优点是高效率、高活力和自动化,缺点是高成本、有限通量;
- 液滴法:优点是通量高,缺点是存在大量未使用的液滴,导致试剂浪费和细胞损失;
- 微孔法:优点是不需要泵或者微流控装置,但是需要有限稀释降低了微孔的利用率;
- 介电泳法:优点是突破了泊松分布,但是芯片制造复杂,通量有限,分离时间长,可能会导致细胞活性低。
基于微孔的scRNA-seq由于其开放环境,可能会在细胞裂解时发生mRNA损失或者交叉污染,目前已有Seq-Well采用了半透膜进行辅助,但操作繁琐。此外还有无细胞mRNA噪声的问题。
作者提出了一种高通量、低成本的Well-Paired-Seq,其双孔结构基于局部准静态流体动力学原理,能够高效捕获细胞、细胞/微珠和无细胞mRNA去除,有助于显著减少细胞损失和背景噪声。 此外,提出了一种新颖且简便的细胞裂解策略,通过在矿物油中引入裂解试剂,在封闭环境中进行细胞裂解,有效避免孔之间的交叉污染。
Overview of the Well-Paired-Seq
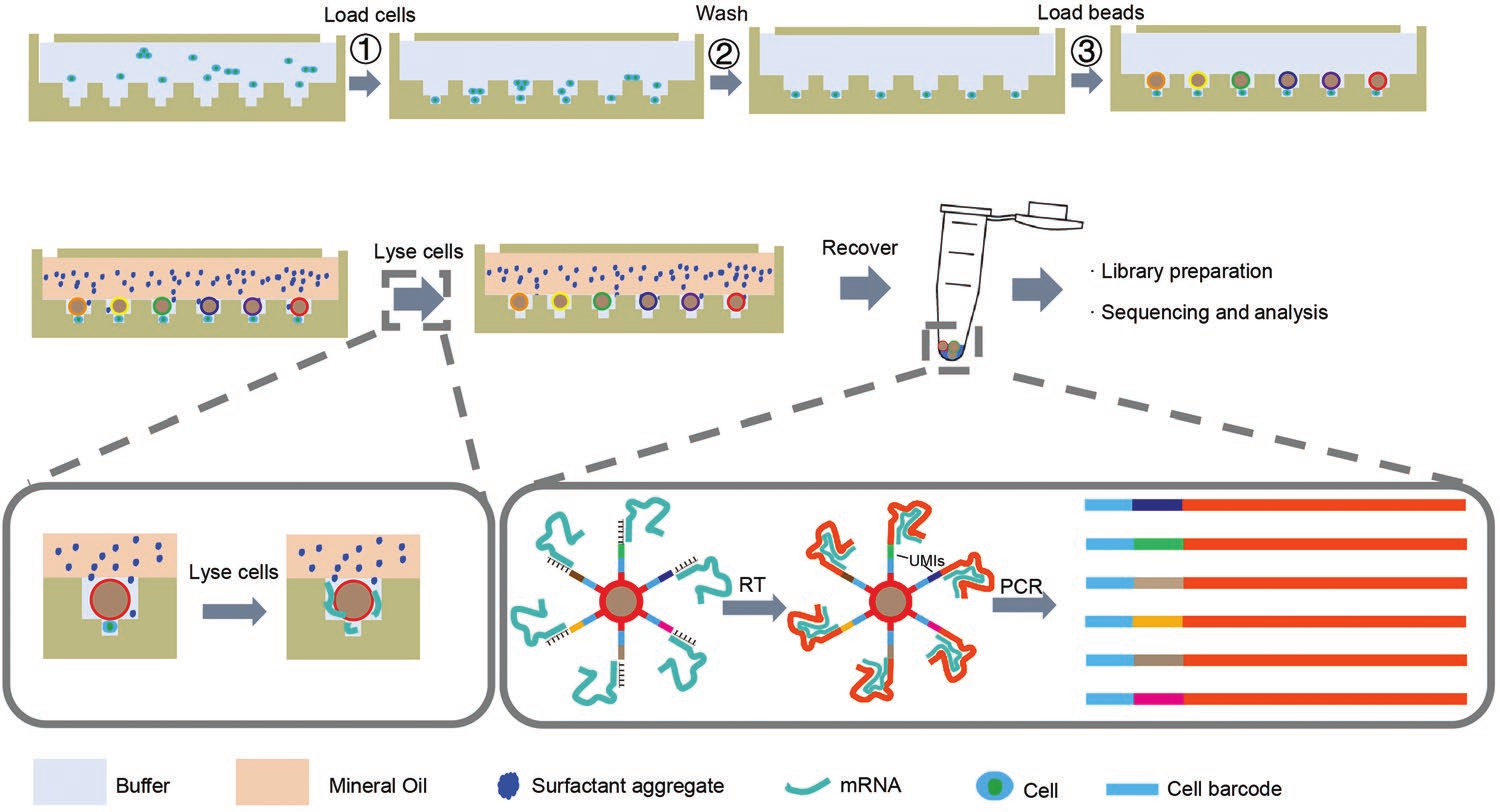
为应对以上挑战,作者发展了一种高通量、高利用率、低成本的单细胞测序新方法:Well-paired-seq,该方法协同尺寸排阻和局部准静态流体动力学原理设计了含数万到数十万个双层微孔的配对芯片,上下层微孔尺寸分别符合编码微球和细胞大小。由于尺寸排阻,每层微孔只能进行单颗粒的沉降捕获,从而实现细胞与微球的一一配对。相对于最上层的流体通道,微孔中流速骤降,最下层的细胞捕获孔可达到准静态,这保证了已捕获的细胞不会因上层流体的扰动而损失,所以通过多次沉降可以实现细胞累积捕获,提高细胞利用率和与微球的配对效率。此外,作者提出了矿物油封闭裂解的新方法,为细胞裂解创造封闭环境,避免孔间mRNA交叉污染。该平台的编码微球上包括通用引物、细胞编码、分子编码和Oligo(dT)30序列。操作流程如下:首先依次捕获细胞和微球,使其一一配对,然后通入含表面活性剂颗粒的矿物油封闭上层微孔,表面活性剂沉降并溶于微孔中的缓冲液,从而对细胞进行裂解。释放的mRNA被微球捕获,回收微球后进行逆转录、cDNA扩增及后续的建库测序,通过识别细胞和分子编码得到高至100, 000个单细胞的基因表达谱。
Structure Design and Characterization of Well-Paired-Seq Chip

- (a)Well-paired-seq芯片的图像。
- (b)SEM图像显示硅片上Well-paired-seq芯片模板结构的俯视图,可见其由数千个位于流道下方的双孔组成。
- (c)Well-paired-seq芯片结构侧视图,设计为底孔和顶孔,用于基于尺寸排阻原理的单细胞/微珠隔离。
- (d)Well-paired-seq芯片中的流体动力学模拟。(i)和(ii)细胞捕获孔设置为20 µm宽和20 µm高。(i)中的三个微珠捕获孔的宽度分别设置为120、85和50 µm(从左到右),高度为50 µm。(ii)中的三个珠子捕获孔分别设置为宽度120、85和50 µm(从左到右),高度为25 µm。
- (e)不同位置的速度如(d)所示,可见条件c下的细胞可视为准静态。此时,当在流动通道中引入较大的流体扰动时,最下层的细胞捕获孔扰动极小,从而实现了在不影响已捕获细胞的情况下,去除微球捕获孔中多余细胞。此外,未捕获的细胞可以通过吹打重悬,进行多次沉降捕获,从而产生累积捕获效应。结构上的尺寸排斥和准静态流体力学原理的结合,理论上可以保证微孔中单个细胞/微珠的捕获,并提高细胞与微珠配对效率。
Single Cells/Beads Pairing, Lysis, and Recovery
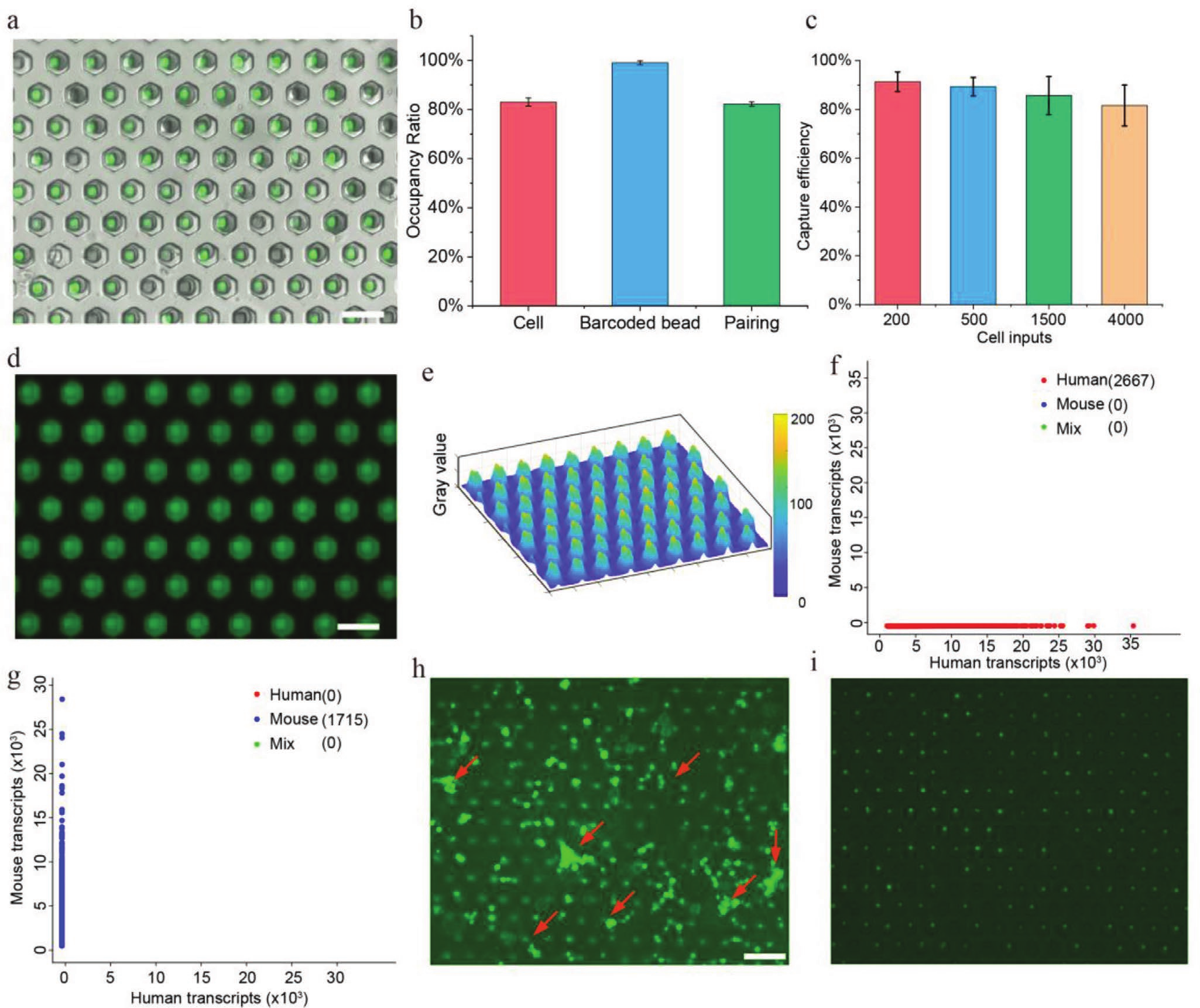
- (a)包含成对单细胞(NIH/3T3)和微珠的Well-paired-seq芯片的图像。
- (b)微珠与细胞占有率及配对率的统计图。微孔的占有率分别能达到83%(细胞)和99%(微珠),配对率约为82%。
- (c)用不同数量的输入细胞评估单细胞捕获效率。通过三次累积捕获均可达到90%左右的细胞捕获率。
- (d)由矿物油密封的双孔中的荧光图像,在芯片中通入荧光染料并用矿物油覆盖。
- (e)显示(d)中Well-paired-seq芯片中荧光强度的3D曲面,与双孔处对比,孔间位置的荧光强度急剧下降,表明矿物油有效的密封了微孔。
- (f-g)在Well-paired-seq中检测无细胞mRNA污染的测序结果。 从人或小鼠细胞中提取RNA(0.7 ng/µL)与另一物种的细胞混合,细胞被捕获至芯片后用缓冲液清洗去除游离RNA并进行后续操作,测序结果表明没有检测到来自其他物种的游离RNA,表明Well-paired-seq芯片具有优越的游离RNA去除能力。
- (h)荧光细胞图像,包含加载在Well-paired-seq芯片中的细胞簇。红色箭头表示细胞簇。
- (i)洗涤未捕获的细胞(包括细胞簇)后分离的单个细胞的图像。表明可以轻松去除细胞悬液样品中的细胞簇,提供高质量数据。以上数据表明,Well-paired-seq细胞利用率和配对率高,具有强大的游离RNA和细胞簇去除能力,减少了下游的数据分析的技术差异和背景噪音。
Cell-Free RNA and Aggregated Cells Removal Capability

- (a)TAMRA荧光染料溶液(红)和NIH/3T3细胞混合物加载到Well-paired-seq芯片中。
- (b)Well-paired-seq芯片在1x PBS洗涤后的荧光图像。
Technical Validation with Cell Lines in Well-Paired-Seq

- (a)加载了单细胞的Well-paired-seq芯片的图像。NIH/3T3细胞(绿色)被DiO荧光染料染色; K562细胞(红色)被DiL荧光染料染色。
- (b)使用Well-paired-seq的人鼠混合实验。平台生成了包括2810个人类细胞和2254个小鼠细胞的高质量文库,测序数据中平均约70% reads比对到外显子区域。
- (c)在芯片中观察到的多细胞率,并分别通过(a)和(b)的测序估计。 测序结果展示的多胞率与芯片上的成像结果相近,低于3.5%。
- (d)评估Well-paired-seq中的捕获率和回收率。 结果表明达到了约为65%的高细胞回收率。
- (e-f)比较不同平台的(e)基因数(6085、5840、6712、5841个基因)和(f)转录本数(39112、27596、32362、23641个转录本)。为评估该平台的灵敏度,对该平台NIH/3T3细胞的测序数据进行了下采样,以匹配读取深度。结果表明Well-paired-seq的转录本和基因检测水平与其他基于编码微球的scRNA-seq方法相当。
- (g-h)不同平台的(g)测定通量和(h)Well-paired-seq不同通量多胞率的比较。通过改变芯片中的微孔数量(10000到200000),该平台的通量在保证低多胞率的情况下(0-6.2%)成功覆盖了4个数量级,检测了70到116101范围的细胞,这体现了Well-paired-seq平台在处理不同通量需求的样品时,具有高的灵活性。
- (i)Well-paired-seq检测到的稀有细胞。通过将老鼠细胞按1:300的比例添加到人类细胞中,测序结果的两物种比例与预期相符(1:350),表明Well-paired-seq平台可以实现对稀有单细胞的无偏倚检测。
Cell Type Profiling Using Well-Paired-Seq

- (a) 五种人肺腺癌细胞系的UMAP图(合计10332个细胞,包括4328个H2228细胞、2626个HCC827细胞、1706个H1975细胞、929个H838细胞和743个A549细胞)。
- (b)UMAP和小提琴图显示了五种人肺腺癌细胞系的基因表达。
- (c)Well-paired-seq检测到的每种细胞类型的细胞比例。两个重复实验中的各细胞占比与原始输入的样本保持高度一致。
- (d)来自按细胞类型着色的外周血单核细胞(peripheral blood mononuclear cells,PBMCs)的单细胞轮廓(点)的UMAP。
- (e)PBMCs单细胞转录组数据中每种细胞类型的点图可视化。点的大小编码细胞类型中细胞的百分比,颜色编码平均表达水平。在检测的所有PBMC细胞中含33.4%的CD4+T细胞,15.8%的CD8+T细胞,23.5%的NK细胞,8.0%的B细胞,13.1%的CD14+单核细胞,4.3%的FCGR3A+单核细胞和1.9%的树突状细胞。以上数据证明了Well-paired-seq在复杂样本中的细胞类型分析能力。
Multi-Sample Parallel Processing Using Well-Paired-Seq

- (a)多通道Well-paired-seq芯片示意图。
- (b)多通道Well-paired-seq芯片的图像。
- (c)UMAP显示重复实验。不同的实验组用不同的颜色标记,结果证明了重复实验之间没有批次效应。
- (d)UMAP显示来自不同SAHA浓度处理的28821个A549细胞簇。
- (e)用不同SAHA浓度处理的细胞映射到(d)中的每个簇上。
- (f)条形图显示(e)中每个簇上的细胞比例。结果表明细胞被分为四个簇并以剂量依赖性方式分布,低剂量处理的细胞显示出较小的差异,而大多数高剂量处理的细胞位于相应的簇3和4中,表明剂量诱导程度更高。然而,高剂量药物处理的一些细胞仍位于簇1和2中,表明对该药物的异质反应。
- (g)四个簇的基因本体富集的生物过程分析。
- (h)显示4个簇细胞中差异表达基因的热图,主要与细胞周期停滞和细胞代谢相关。
- (i)每个簇上的增殖指数映射,表示作为G1/S期或G2/M期标记的转录物的聚合表达水平。
- (j)不同SAHA浓度处理的细胞的剂量依赖性轨迹。总之,这些结果表明Well-paired-seq能够并行处理样本并提供单细胞的综合基因表达分析,以揭示药物反应的异质性。
SAHA可以抑制HDAC(组蛋白去乙酰化酶)活性,它具有多种细胞效应,包括乙酰化组蛋白的积累和细胞周期停滞。
Discussion
发展了一种新的单细胞测序方法:Well-paired-seq:
- Well-paired-seq协同尺寸排阻和局部准静态动力学原理,在细胞捕获、细胞/微球配对、游离mRNA去除等方面具有出色的效率,显著地降低了细胞的损失和背景噪音,以较低的双胞率实现了跨越4个数量级(100-100000)的细胞高通量分析。
- 将整个捕获过程集成到芯片上,无需外围设备,操作简单。
- 提出的矿物油封闭裂解策略有效避免了微孔间的交叉污染。
- 利用Well-paired-seq结合高通量测序,全面分析了人PBMCs和药物处理细胞的转录表达,揭示了单细胞的异质性。
Reference
Yin K, Zhao M, Lin L, et al. Well-Paired-Seq: A Size-Exclusion and Locally Quasi-Static Hydrodynamic Microwell Chip for Single-Cell RNA-Seq[J]. Small Methods, 2022, 6(7): 2200341.