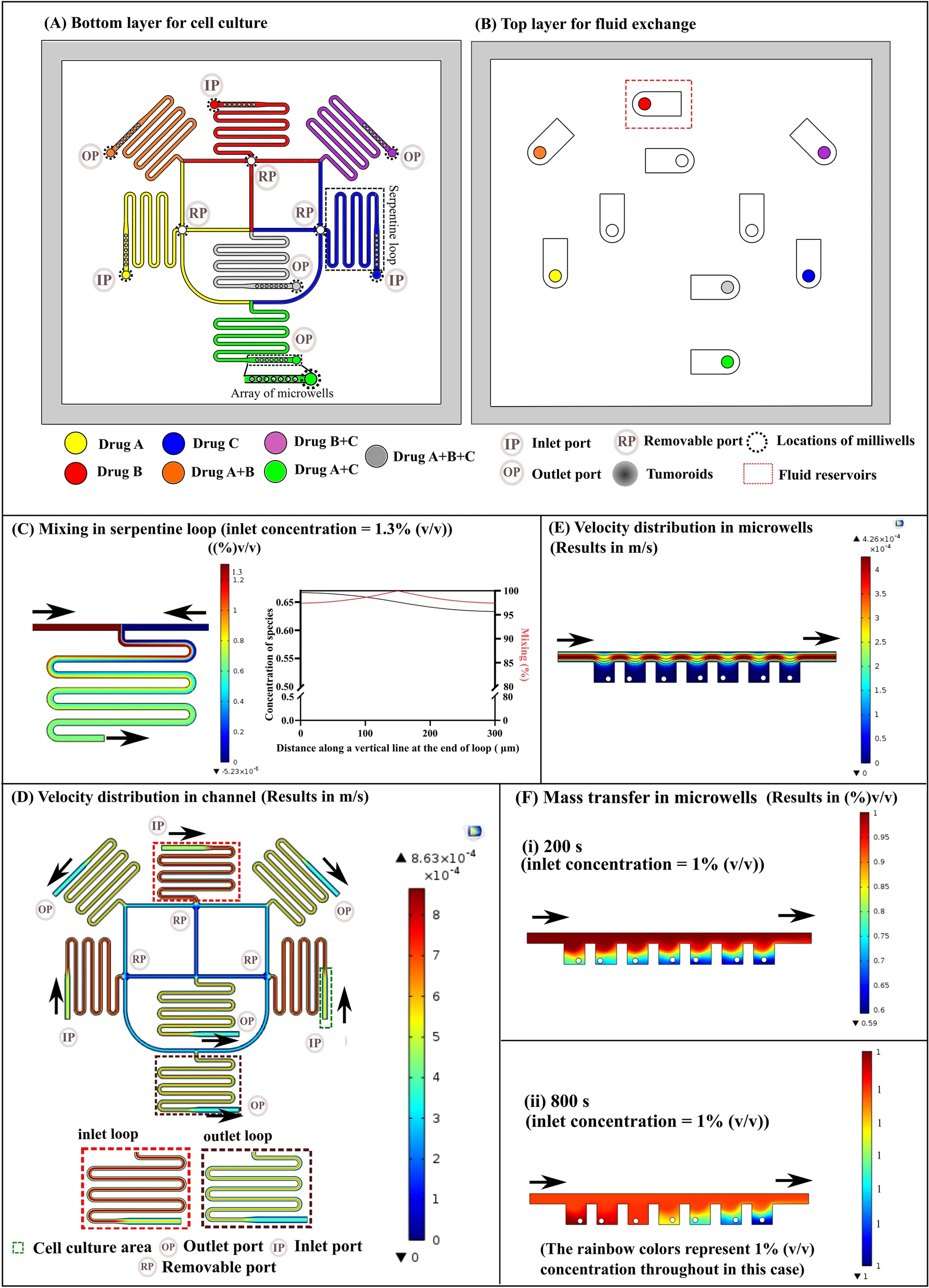
一种可拉伸的高通量三维细胞微阵列平台,可以对封装在三维微凝胶中的细胞施加动态机械应变,筛选细胞反应。
Introduction
思路:
- 体内的细胞不断受到多种微环境的刺激,包括生化信号、生物力场、细胞外基质(ECM)或基质硬度以及细胞间的相互作用。他们共同(线索之间存在相互作用)决定了细胞的命运、功能和反应。因此,评估细胞对多种微环境线索协同作用的反应将受益于组合和高通量(HT)方法。
- 现在的细胞芯片平台大多主要集中在筛选可溶性生化因子和ECM蛋白的影响,只有少数平台可以筛选机械刺激,而体内的细胞通过机械传导过程感知和响应各种机械刺激。
- 一些用于筛选细胞机械反应的细胞微阵列平台已经被开发出来。然而因为细胞在三维中的行为与二维中的行为完全不同,所以应该开发三维的平台。目前只有少数平台展示了筛选3D细胞对机械刺激反应的能力。
- 在本文中报告了一个可拉伸的三维细胞微阵列平台,其制造步骤简单,吞吐能力提高。所开发的平台由粘合在弹性复合膜基底上的三维生物打印的纤维细胞填充的GelMA微凝胶阵列组成。使用定制的单轴动态拉伸器对该平台进行动态单轴拉伸,以调查细胞对机械应变的反应。
Fabrication of Stretchable 3D Cellular Microarray Platform
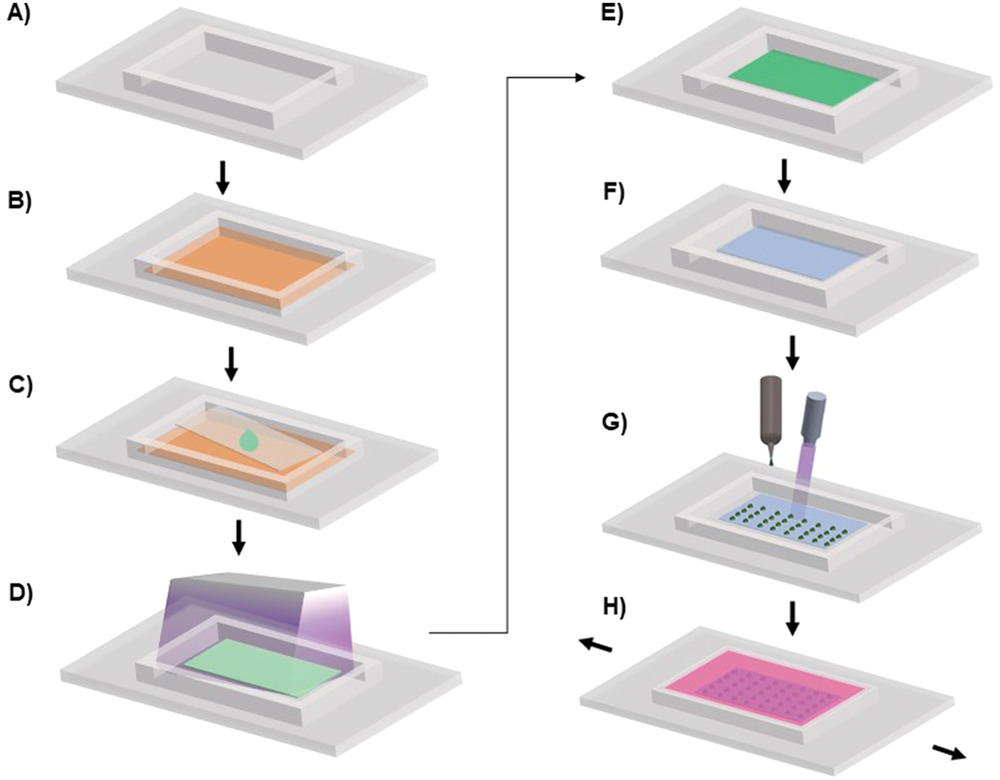
- (A)制造一个PDMS膜(厚度为750 μm)。由于GelMA已知与PAAm结合,而其本身并不具有与PDMS结合的亲和力,因此需要将PAAm整合到PDMS膜上。
- (B)二苯甲酮处理PDMS膜表面。具体而言,将10% w/v的二苯甲酮溶液溶解在丙酮/水混合物 (65:35 w/w) 中,然后浸泡。丙酮使PDMS网络膨胀,从而使二苯甲酮吸收到PDMS膜的表面,以减轻PDMS的氧抑制作用。
- (C)将预聚物聚丙烯酰胺(PAAm)溶液分配到吸收二苯甲酮的PDMS表面上,并用玻璃盖板覆盖。
- (D)将整个装置暴露在紫外线(UV)下。在此期间,二苯甲酮(一种紫外线反应性分子)促进了PAAm在PDMS表面的光移植和聚合。
- (E)形成一个PAAm凝胶垫。
- (F)在PAAm/PDMS膜复合基底的表面涂上一薄层FluoroPel。由于PAAm亲水,所以用FluoroPel(一种氟丙烯酸酯聚合物)旋涂在基底的PAAm垫上形成一个薄的超疏水层。
- (G)将含有细胞的GelMA预聚物溶液进行喷墨生物打印并同时交联(蓝色激光二极管交联),在复合膜基底上形成GelMA微凝胶阵列。
- (H)可拉伸的三维细胞微阵列平台最终暴露在动态机械刺激下。
Characterization of Stretchable 3D Cellular Microarray Platform

- (A)复合基底的PAAm垫的表面润湿性能的变化分析。
- (i, ii)水合情况下,未处理(i)和Fluoropel处理基底(ii)GelMA液滴水接触角;
- (iii, iv)脱水条件下,未处理(iii)和Fluoropel处理基底(iv)GelMA液滴水接触角;
- (v)GelMA在不同基底条件下的接触角的统计分析。结果表明,超疏水的Fluoropel涂层有助于在复合基底上形成定义明确的GelMA液滴的三维结构。
- (B)GelMA粘合测试。将GelMA液滴分配并交联在:
- (i)涂有Fluoropel的复合基底;
- (ii)涂有Fluoropel的PDMS基底;
- (iii)当暴露在涡流条件下时,GelMA水凝胶点与复合基底强烈结合;
- (iv)同样地涡流条件,PDMS基底不能与GelMA水凝胶结合。结果证实,Fluoropel本身在GelMA与基底的粘合中没有作用,而PAAm是介导GelMA与复合基材永久粘合的必要条件。

- (A) 定制的生物打印机装置。增加一个环境室来控制温度和湿度,避免凝胶凝固或者GelMA微滴蒸发。
- (B)打印加载了绿色染料的GelMA预聚物溶液,同时使用激光二极管交联,形成微凝胶阵列。
- (C) 打印的GelMA微凝胶阵列的俯视图,它与复合膜基底结合在一起。
- (D)侧视图显示了打印的GelMA微凝胶的明确的三维结构。

- (A) 一个定制的、可安装在显微镜上的、单轴细胞拉伸装置。
- (B)将打印在复合膜基底上的充满细胞的GelMA微凝胶阵列:
- (i)连接到拉伸装置上;
- (ii)以10%的应变进行拉伸。(黑色箭头表示拉伸方向)。
- (C)记录GelMA微凝胶在非拉伸和拉伸条件下的显微图像,并计算颗粒之间的位移来测量应变(比例尺呈现50 μm,白色箭头表示拉伸方向)。
- (D)应变传递分析显示,当以10%的应变拉伸时,超过90%的应变从PDMS膜传递到印在整个PAAm凝胶垫上的GelMA微凝胶(膜基底的左端、中间和右端)。这一结果验证了封装在印刷的GelMA微凝胶中的细胞也能感受到施加的应变。
- (E)计算模拟显示了膜以10%的应变率单轴拉伸时的应变分布:
- (i)从拉伸的PDMS膜到GelMA微阵列的应变转移;
- (ii)拉伸的6%GelMA微阵列的单个GelMA微凝胶中的应变梯度形成,拉伸应变在整个微凝胶体积内的分布不均,微凝胶在其底面附近经历了最大的应变,以其在那它与PAAm垫表面接触。
- (iii)拉伸的6%GelMA微阵列的单个GelMA微凝胶内的应变场向量。
Biocompatibility of Stretchable 3D Cellular Microarray Platform

- (A)15×5细胞的GelMA微凝胶阵列打印后的活/死分析。
- (B)印刷后的一个代表性满载细胞的GelMA微凝胶。
- (C)显示三维GelMA微凝胶内细胞分布的三维等轴视图。
- (D)第0 d和第7 d的细胞活力分析显示,在生物打印过程后,甚至在培养箱内培养7 d后,细胞的活力超过90%。
- (E)第7 d,细胞核(DAPI)/F-肌动蛋白(Phalloidin)荧光染色的微凝胶阵列显示了成纤维细胞在打印的GelMA微凝胶阵列中的附着、延伸和三维细胞网络的形成。
- (F)第0 d和第7 d的细胞数显示了3D GelMA微凝胶内培养细胞的增殖情况。
HT Analysis of Cell Mechano-Responses

- (A)细胞核(DAPI)/F-肌动蛋白(Phalloidin)荧光染色的拉伸和非拉伸(对照)细胞装载的3D GelMA微凝胶阵列的代表性凝胶(白色箭头表示拉伸方向)的正交投影(i)、俯视(ii)和3D等距视图(iii)。
- (B)在拉伸复合膜基底的不同位置(左端、中心和右端)的拉伸和对照微凝胶阵列的细胞核/F-肌动蛋白染色的GelMA微凝胶(白色箭头表示拉伸方向)。存在于GelMA核心区域的细胞被认为是朝着拉伸方向排列的。此外,暴露在机械刺激下的细胞的肌动蛋白丝的外观与对照组的细胞相比显得更细长。
- (C)细胞排列分析显示在拉伸和对照的三维GelMA微凝胶阵列内被包裹的细胞核在-90°到90°的各个角度的方向。其中,方向接近0°的细胞的细胞核被认为是朝向循环拉伸的方向排列。
Mechanical Properties of GelMA Hydrogels
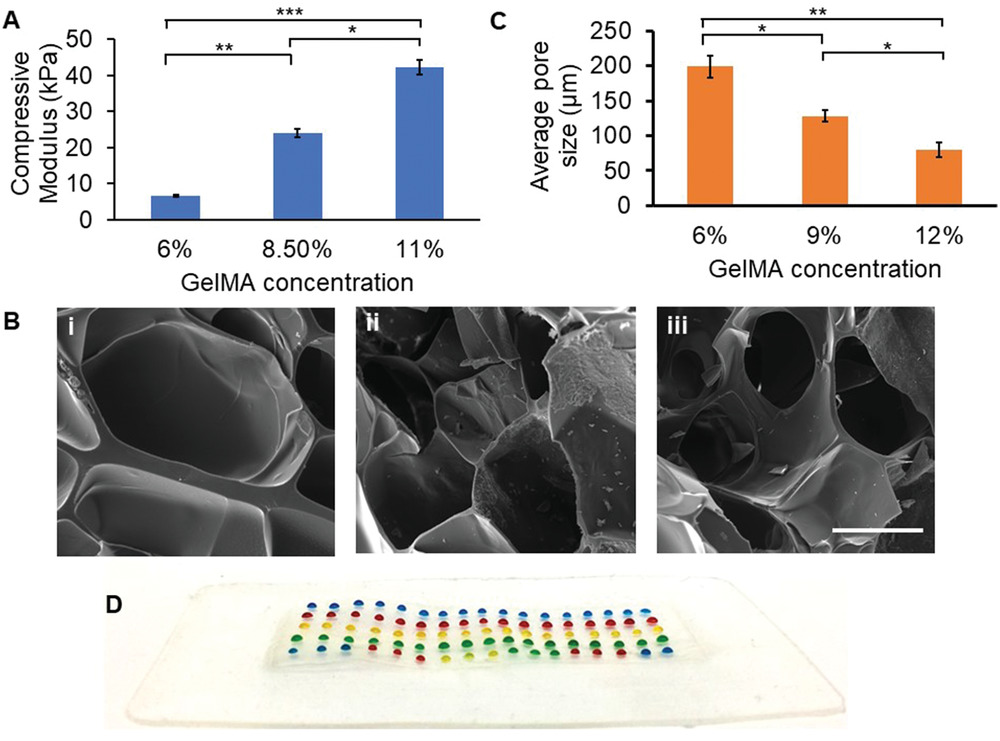
- (A)不同浓度的GelMA-6%、8.5%和11%的压缩模量。低硬度的GelMA水凝胶被证明有利于高水平的细胞增殖,只有在机械刺激下,细胞才能在所有三种浓度的水凝胶(6%、8.5%和11% GelMAs)中扩散和增殖。
- (B) 6%(i)、8.5%(ii)和11%(iii)的GelMA的扫描电子显微镜图像。
- (C)不同浓度(6%、8.5%和11%)的GelMA的平均孔径。GelMA的浓度越低,交联水凝胶中可用的聚合物网络就越少,这就导致了更大的孔隙尺寸。
- (D) 多材料打印能力。在开发的膜复合基底上,以组合的方式对加载不同颜色(对应不同浓度GelMA)的GelMA微凝胶进行生物打印。
Combinatorial Analysis of Cell Mechano-Responses
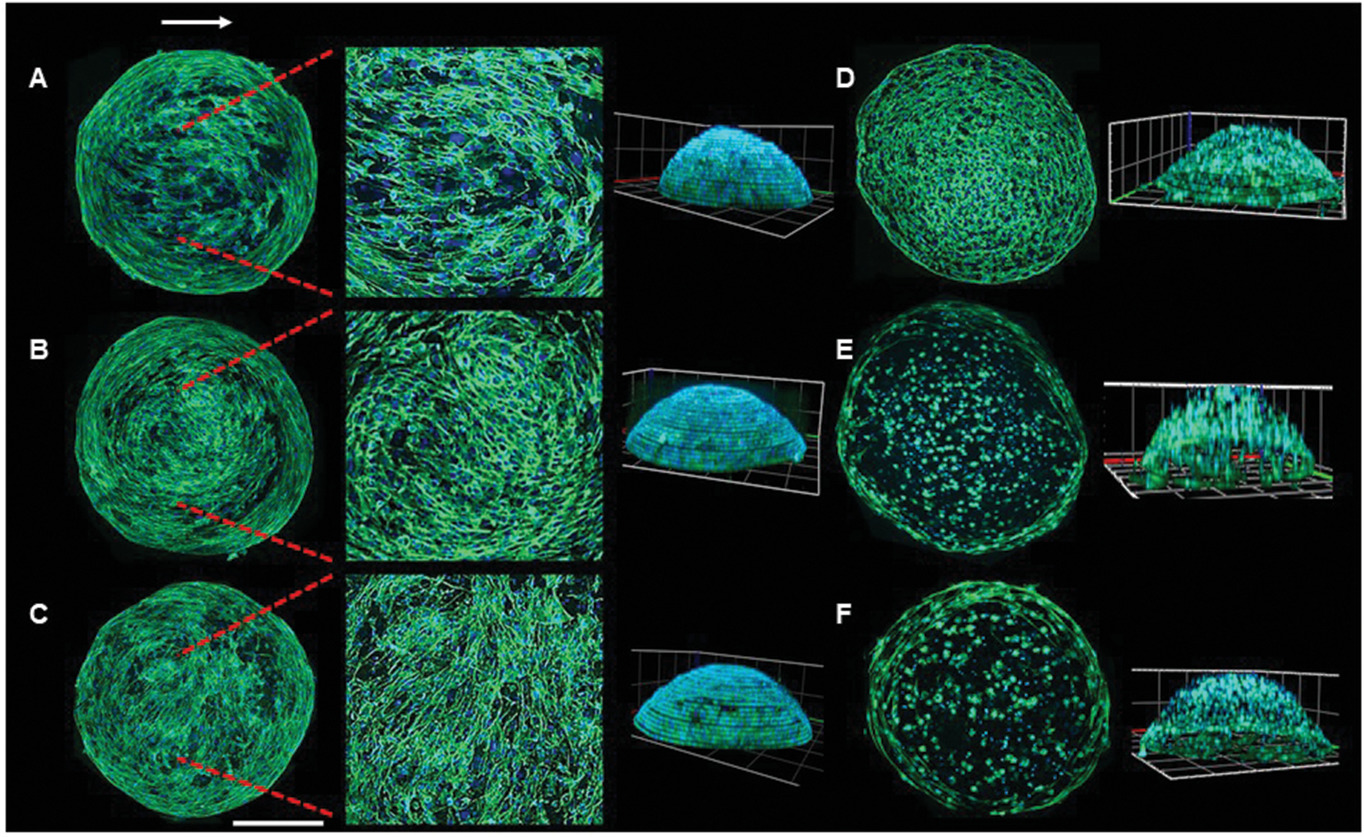
- (A-F)不同硬度的GelMA水凝胶微环境中细胞机械反应的组合筛选:
- (A)6%的拉伸组;
- (B)6%的对照组,可以发现6%的GelMA微凝胶的核心区域的细胞平行于拉伸轴排列;
- (C)8.5%的拉伸组;
- (D)8.5%的对照组;
- (E)11%的拉伸组;
- (F)11%的对照组,与之前的试验类似,细胞在6%的GelMA微凝胶的核心和周边区域显示出随机和周向排列。然而,在8.5%和11%的GelMA微凝胶的核心区域的大多数细胞没有伸长,仍然是球形的,而外围区域的细胞则伸长并周向排列。
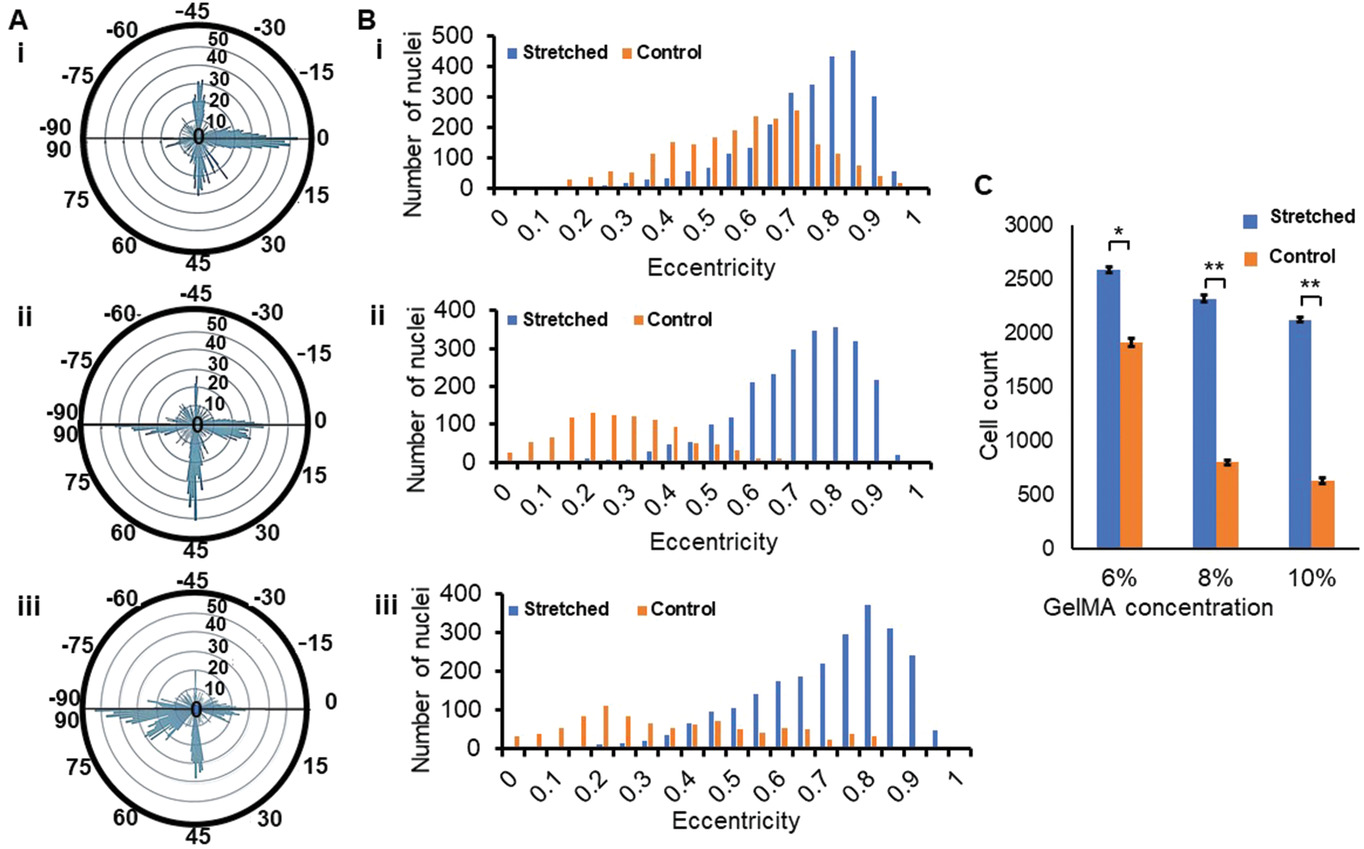
- (A)被包裹在拉伸组中的细胞排列分析,分别为 6%(i)、8.5%(ii)和11%(iii)的GelMA微凝胶阵列。细胞核方向分析的结果验证了作者以前对6%、8.5%和11%的GelMA微凝胶内的平行、混合和垂直细胞排列的观察。
- (B)拉伸组和对照组内封装的细胞的细胞核偏心率分析,分别为 6%(i)、8.5%(ii)和11%(iii)的GelMA微凝胶阵列。玫瑰图描述了从-90°到90°每个角度的细胞核数量。表明细胞在所有三种浓度的GelMA微凝胶中都是高度拉长的。
- (C)拉伸组和对照组内细胞数量的比较。与对照组相比,拉伸组的细胞核数量明显较多。
Discussion
相对于刚性的玻璃载玻片基底,基于PDMS的基底是为充满细胞的三维水凝胶引入机械应变的更好选择。在这项研究中,作者提出将充满细胞的水凝胶微阵列打印在一个弹性材料基底上,可以以HT和组合的方式应用动态机械拉伸。
Hydrophobic Surface Modification of PAAm Gel Pad
复合膜基底的PAAm垫是高度亲水的,试图分配GelMA预聚物溶液时,它就会润湿表面,不会形成一个明确的三维形状。因此需要用一种合适的试剂来改变PAAm垫的表面润湿性,使其具有疏水性。FluoroPel,一种氟丙烯酸剂,通常用于形成薄的超疏水层,用于电泳设备。作者将FluoroPel溶液旋涂在PAAm上,成功地改变了其表面的高疏水性。
PAAm-GelMA Bonding for Applied Strain Transfer
打印的GelMA微阵列必须与复合基底紧密结合,这样当PDMS膜被拉伸时,施加的应变会转移到GelMA微凝胶上。如前所述,GelMA本身就与PAAm形成结合,FluoroPel涂层的存在应该不会影响它们的结合能力。当作者测试GelMA与涂有FluoroPel的PAAm垫子的粘附性时,尽管将其暴露在恶劣的条件下(如高涡流、在液体介质中拉伸多日、浊水流动),它仍然与复合膜基底紧密结合。在随后的实验中发现,超过90%的应变是由被拉伸的基底传递给GelMA的。
Mechanisms of Cell Alignment in Response to Dynamic Strain
一些细胞类型,特别是收缩性细胞(包括成纤维细胞、平滑肌细胞、心肌细胞和人类间充质干细胞(hMSCs)),已经显示出对外部动态刺激的反应。一般来说,当暴露于静态或动态拉伸时,嵌入三维水凝胶的细胞会与拉伸轴对齐。 当该平台以10%的应变率、1Hz的频率拉伸3 d时,6%的GelMA微阵列内的细胞也向拉伸方向平行排列,这一结果与现有文献很一致。
Cell Alignment at Single Cellular Level
最初,细胞通过接触引导来拉长和排列,这种现象使细胞和肌动蛋白丝(细胞内的应力纤维)的方向受基质的几何线索或基质内的纤维(在三维水凝胶/电纺纤维的情况下)的支配。ECM纤维最初与循环拉伸方向对齐,通过接触引导机制促进细胞沿着纤维的方向排列。以前的研究表明,软质胶原纤维倾向于沿着施加应变的方向排列。因此,当外部应变施加到GelMA微凝胶上时,微凝胶纤维可能已经朝着拉伸方向排列,这反过来可能会支配细胞朝着类似的方向排列。由于交联的微凝胶阵列中的GelMA纤维最初是随机的,没有暴露在任何动态的机械拉伸条件下,细胞排列也是随机的,这也是由于接触引导现象造成的。使用SEM可以看到纤维排列。
在三维水凝胶中,细胞不断改变它们的形状,并与它们的微环境进行机械性互动,以迁移或对外部力量作出反应。细胞与微环境相互作用的这一动态过程被称为焦点粘附(FA)成熟,细胞不断重塑其细胞骨架、周围的ECM网络,并通过FA(细胞机械传导的主要枢纽)与ECM连接。一般来说,细胞会对周围的水凝胶纤维网络施加收缩力,并在伸长时招募纤维。收缩力在基质内引起局部张力,与细胞收缩力相平衡,从而保持平衡。当外部动态应变施加到充满细胞的水凝胶上时,会在整个水凝胶中产生一个整体应变,从而导致纤维中张力的增加。为了保持系统的平衡,细胞通过肌动蛋白收缩过程增加其收缩力。细胞收缩力的增加增加了细胞的细胞内张力,并产生应力纤维束(肌动蛋白丝),促进其FA成熟和形成向拉伸方向的延伸/假脚。
6%的GelMA微阵列的杨氏模量为6.6 kPa,这通常被认为是一种顺应性水凝胶。由于成纤维细胞的性质是高度收缩的,它促进了FA的成熟,并沿拉伸方向产生了大量的应力纤维。它还会产生一个高的局部横向压实应变(由于6%的GelMA的顺应性),其幅度大于应用的应变。一方面避免了局部应变,另一方面FA成熟过程的增加导致6% GelMA微阵列中的成纤维细胞平行于拉伸方向重新定位。
Cell Alignment at Multicellular Level
上述理论描述了单个成纤维细胞如何沿拉伸方向排列。然而,在多细胞水平上的细胞排列分为三个阶段。最初,单个细胞平行于拉伸方向排列,然后是细胞在平行方向上相互排列。随后,细胞相互联合,形成一个平行于拉伸轴的弦状结构。细胞的强烈联合是高收缩性、FA成熟和避免应变的结果。弦状结构使细胞显得更加修长,这可能是我们观察到6%拉伸GelMA微凝胶阵列中封装的成纤维细胞比6%对照GelMA微阵列中随机排列的细胞修长程度高的原因。另一个重要的观察结果是,弦状结构主要见于比较浓度较低的GelMA微凝胶。这是因为当细胞密度较高时,细胞与细胞之间的粘附在各个方向上发生的几率很高,阻止了清晰的串状结构的形成。 当细胞与细胞之间的粘附较强时,细胞就有可能形成网状的图案。因此,在未来,在生物打印过程中控制细胞密度可以改善整个拉伸的GelMA微阵列的弦状结构的形成。
Cell Migration Ability within the Aligned vs Random Hydrogel Fibers
细胞与拉伸方向对齐需要细胞在水凝胶内迁移。因此,细胞迁移是使细胞向施加的应变方向重新定向的主要因素之一。三维水凝胶内的细胞迁移可以用吊索短距离迁移(SSM)理论来解释。如前所述,成纤维细胞通过接触引导机制,沿着水凝胶纤维突出它们的延伸和极化。在这个过程中,细胞发生肌动蛋白收缩,并招募附近的水凝胶纤维来储存其弹性应变能量。当施加外部应变时,它超过了纤维的拉力,导致细胞后缘的FA或接触失败。随后,纤维反冲,在此期间,储存的弹性能量被释放并转移到细胞中,使其向拉伸方向运动。细胞的SSM主要是在排列的纤维而不是随机的纤维中观察到的。因此,细胞的重新定向可能是两种理论的协同作用–水凝胶纤维向拉伸方向排列、避免应变和由于高肌动蛋白收缩性而导致的更强的ECM成熟。
Circumferential Cell Alignment along the Boundary of GelMA Microgels
其他支配细胞重新定向的因素是应变率、频率、应变条件的持续时间和水凝胶的几何形状。从我们的结果来看,只在GelMA微凝胶的核心区域观察到主要的重新定向;然而,细胞沿其边界周向排列。在GelMA微凝胶的边界,几何效应(来自半球形的GelMA微凝胶)可以支配施加的拉伸应变,导致细胞在圆周方向上排列。同样地,在核心区域,拉伸应变主导了几何效应,使细胞平行于应变方向。
Discrete Cell Alignment within Different Stiffness Microenvironments
SSM理论的另一个重要结果是,细胞迁移的程度高度依赖于水凝胶基质的硬度。同时,水凝胶的硬度会影响水凝胶内的水凝胶纤维招募、细胞收缩性、FA成熟度和横向压实应变的水平,这就提出了一个问题:当细胞被包裹在不同硬度的微环境中并被循环拉伸时,它们会有什么反应。为了检查水凝胶微环境硬度和循环应变的组合效应,作者将6%、8.5%和11%的GelMA微凝胶印在同一个复合膜基底上,然后在1Hz频率下承受10%的应变,持续3 d。
基于上述假设预计会观察到细胞排列趋势的差异。正如预测的那样,观察到随着GelMA浓度的增加,水凝胶的机械强度增加,孔隙大小减少。无论GelMA的浓度和硬度如何,几何效应在微凝胶的外围区域占主导地位,因此在所有三种情况下,细胞都在圆周方向上排列。然而,我们观察到在6%、8.5%和11%的拉伸细胞填充的GelMA微阵列的核心区域,细胞方向分别是平行、混合和垂直的。细胞取向的这些差异可以根据上述假设再次解释:随着GelMA硬度的增加,细胞的收缩力不足以招募和拉伸较硬的纤维。因此,由于细胞的收缩力较小,纤维中存在的拉力也较小,这就大大降低了微凝胶内局部的横向压实应变。同时,较硬的GelMA不会发生大量的变形,导致沿外部施加的应变方向的压应力和应变的发展。在8.5%(中等硬度)和11%的GelMA(高硬度)微凝胶的情况下,沿拉伸方向增加的压应力和应变会导致细胞避开拉伸方向,从而在混合和垂直方向上排列。
细胞迁移能力也受水凝胶硬度的影响。根据SSM理论,与较硬的水凝胶相比,较软的水凝胶(6 kPa)中的细胞发生迁移的速度要快5倍(6%的GelMA硬度为6.6 kPa)。较硬的纤维中缺乏拉力,减少了储存的弹性能量,这又降低了细胞迁移率。作者还模拟了当复合膜以10%的应变拉伸时,具有不同压缩杨氏模量的GelMA微凝胶中的应力分布曲线。随着杨氏模量的增加,观察到沿拉伸方向的应力水平增加,而且在微凝胶表面的径向方向也是如此。因此,模拟结果与作者对细胞排列的假设一致。
除了细胞重新定向外,循环拉伸使细胞形成高度接近体内条件的结构,也提升了它们的分化能力和功能。这可能是观察到大量细胞在坚硬的水凝胶中伸长和重新定向的原因(拉伸时),这表明细胞正在经历应变。然而,在非应变的较硬的GelMA微凝胶(8.5%和11%)中的细胞并没有伸长很多。这些结果也与现有文献一致,表明细胞在较硬的三维水凝胶中没有附着、伸长和增殖。一种可能的解释是由于溶解因子的扩散速度受到较硬水凝胶中较小孔径的影响。因此,较硬的非拉伸微凝胶外围的细胞似乎会伸长,因为它们有最大的扩散。细胞保持圆形的另一个原因可能是由于它们无法降解较硬的微凝胶基质。另外,招募僵硬的微凝胶纤维对细胞来说是难以附着和伸长的。然而,作者推测动态刺激(在我们的案例中是循环拉伸)增强了细胞在较硬的水凝胶内的扩散,这与其他类似的研究是一致的。进一步调查细胞的功能,如用拉伸的和控制细胞的GelMA微凝胶阵列的机械传导和细胞间信号,将有助于确定水凝胶微环境属性在机械刺激下对细胞行为的影响。
Outlook
这里介绍的平台具有高度的通用性,可用于多种机械生物学研究。作为概念验证,作者开发了一个三维细胞微阵列平台,用于对细胞进行机械刺激,并以HT和组合方式表征其反应。主要筛选因素是细胞扩散、增殖,以及暴露在动态机械刺激下时与生物材料的相互作用。研究结果证明,细胞以高通量方式经历应变,可以在整个可拉伸的微凝胶阵列中实现不同的细胞排列。
各种类型的细胞也可以在同一基底上形成图案,特别是使用挤压或SLA生物打印技术,并可以监测它们的联合细胞反应,这可以以HT方式提供对3D共培养细胞的反应的洞察力。同时平台可以扩大规模,引入广泛的其他微环境线索,并以HT方式筛选细胞反应和3D组织的形成。引入多种线索对干细胞分化研究极为重要,可以分析应变、微环境硬度、几何效应和生物化学刺激效应的综合影响。
平台的另一个缺点是,它只允许细胞暴露在单一的应变水平上。为了用相同的复合膜基底实现应变梯度,它的形状可以改变,这样细胞就可以根据它们在基质上的位置受到不同程度的机械刺激而产生应变。此外可以修改基底的设计,使其在培养室中有多个独立的隔间,从而形成一个可拉伸的微孔阵列。这种可拉伸的微孔阵列有可能被用于心脏/肌肉骨骼疾病的药物和生长因子的HT筛选,以及干细胞分化。
除了平台,该单轴拉伸器设备可以安装在显微镜上,用它可以捕捉到细胞对外部机械刺激的反应的延时图像,这将更好地描述细胞如何对准拉伸方向。细胞培养基也可以很容易地通过单轴拉伸器的夹持单元的延伸部分进行灌注,这将允许细胞在暴露于外部机械刺激时进行动态培养。
Conclusions
作者展示了可拉伸的细胞微阵列平台,用于动态拉伸的三维细胞反应的HT分析。该平台是由PDMS膜与FluoroPel涂层的PAAm凝胶垫粘合而成的。喷墨打印系统被用来打印充满细胞的GelMA微凝胶阵列。所开发的平台显示出高度的生物相容性,以促进细胞在三维GelMA微凝胶中的粘附和增殖。当复合膜基底被拉伸时,它也有利于应变转移到封装在GelMA微阵列中的细胞(从细胞向拉伸方向的重新排列可以看出)。该平台的组合分析能力通过显示不同细胞微环境硬度下不同的细胞排列和扩散行为,以及没有动态机械刺激的情况下,得到了证明。该平台可以扩大规模,引入广泛的其他微环境线索,从而以HT方式筛选细胞反应和三维组织的形成。
Reference
Sakthivel K, Kumar H, Mohamed M G A, et al. High Throughput Screening of Cell Mechanical Response Using a Stretchable 3D Cellular Microarray Platform[J]. Small, 2020, 16(30): 2000941.